 |
ヒアルロン酸(HA)は、ᴅ-グルクロン酸(GlcA)とN-アセチル-ᴅ-グルコサミン(GlcNAc)の二糖繰り返し構造からなる非硫酸化グリサミノグリカンである。HAは脊椎動物において細胞外マトリックスの主要な構成分子であり、Streptococcus pyogenesやPasteurella multocidaなどある種の細菌では、莢膜多糖として宿主免疫機構の回避にはたらく。この多糖の生合成を担う酵素が、HA合成酵素(HAS)である。アミノ酸配列とタンパク質の予測される構造の類似性に基づいて、HASは2つのファミリーに分類される。連鎖球菌やクロレラウィルス、脊椎動物のHASは、クラスI ファミリーに属し、Pasteurella multocidaのpmHASはクラスII ファミリーに属する(1, 2)。クラスIとII HASは、その構造や膜トポロジーおよびHA合成機構が各々異なっている。
クラスI HASは、セルロース合成酵素(EC 2.4.1.12)やキチン合成酵素(EC 2.4.1.16)などと同じglycosyltransferase family 2(GT2ファミリー)に分類される糖転移酵素である(http://www.cazy.org/GT2.html)。クラスI HASは、アミノ末端とカルボキシ末端側に疎水性アミノ酸からなる複数の膜貫通ドメインや膜結合ドメインをもつ(図1)。分子の中央部には、保存されたD、DxD、QxxRWモチーフをもつ触媒部位がある。これらのモチーフは、糖供与体ウリジン二リン酸 (UDP)-GlcNAcとUDP-GlcAから単糖を伸長しているHAに交互に転移するために必要とされる(図1)。クライオ電子顕微鏡解析により明らかとなったクロレラウィルスHAS(Cv-HAS)の立体構造は、膜貫通ドメインが膜に挿入され、触媒部位が細胞質に向いているというトポロジー分析の結果とよく一致する(3)。また、構造解析の結果は、UDP-GlcNAc基質の結合と加水分解、そしてGlcNAcプライミングが、この酵素の膜貫通ドメインのコンフォメーション変化を引き起こし、HA分泌のための新たな膜貫通チャンネルを形成することを示唆している(図2)。Cv-HASは単量体であり、GlcNAcプライマーから非還元末端に向かってHAを伸長するのに対し、細菌のHASは二量体化してHAの還元末端を伸長させることが報告されている(4)。一方、クラスII HASは、カルボキシ末端付近の膜付着ドメインと糖転移酵素活性をもつ2つのドメインから構成され、HA鎖の非還元末端を伸長する(4)。
脊椎動物のHASには、HAS1-3の3種のアイソフォームがあり、それらは活性や生成物の伸長速度、そして安定性などの性質が異なる(5)。哺乳類のHASアイソフォームの中で、HAS2の活性と代謝回転は、リン酸化やO-GlcNAc化、およびユビキチン化などの翻訳後修飾により厳密に制御されている(6)。さらに、天然のアンチセンス転写物HAS2-AS1が、HAS2 mRNAの安定化を制御している(6)。このように、HA合成は、異なるHASの転写や翻訳、翻訳後修飾など多段階で厳密に制御されている。
HAS遺伝子の同定と最近のCv-HASの立体構造の決定は、HA生合成機構に関する新たな知見をもたらしてきた。しかしながら、HA鎖の伸長方向やHA分泌、そしてHAS活性制御に関する多くの課題は、その解明が待たれる。
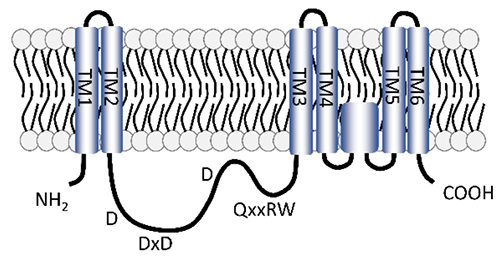
図1.脊椎動物HASの予想される構造
TM1~6は膜貫通ドメインを表し、D、x、R、W はそれぞれアスパラギン酸、いずれかのアミノ酸、アルギニン、トリプトファンを表す。
|
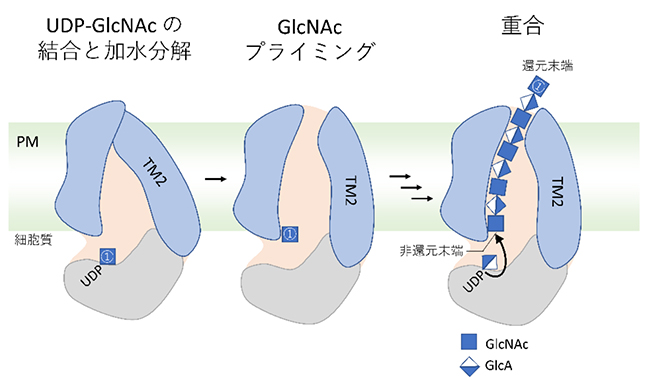
図2.HA伸長と分泌の予測モデル(Cv-HASの例)
UDP-GlcNAc基質の結合と加水分解、そしてGlcNAcプライミングが、膜貫通ドメインのコンフォメーション変化を引き起こし、HA分泌のための膜貫通チャンネルを形成する。GlcNAcプライマーを起点に、UDP-GlcAとUDP-GlcNAcから単糖が交互に転移して、HAが合成される。合成されたHAは、膜貫通チャネル内を通って細胞外へと分泌されると考えられる。GlcNAcプライマーは①で記す。PM:細胞膜、TM2: 膜貫通ドメイン2
|
板野 直樹(京都産業大学生命科学部)
| References |
| (1) |
Weigel PH: Functional characteristics and catalytic mechanisms of the bacterial hyaluronan synthases. IUBMB Life 54, 201-211, 2002 |
| (2) |
Itano N, Kimata K: Mammalian hyaluronan synthases. IUBMB Life 54, 195-199, 2002 |
| (3) |
Maloney FP, Kuklewicz J, Corey RA, Bi Y, Ho R, Mateusiak L, Pardon E, Steyaert J, Stansfeld PJ, Zimmer J: Structure, substrate recognition and initiation of hyaluronan synthase. Nature 604, 195-201, 2022 |
| (4) |
Weigel PH, DeAngelis PL: Hyaluronan synthases: a decade-plus of novel glycosyltransferases. J. Biol. Chem. 282, 36777-36781, 2007 |
| (5) |
Itano N, Sawai T, Yoshida M, Lenas P, Yamada Y, Imagawa M, Shinomura T, Hamaguchi M, Yoshida Y, Ohnuki Y, Miyauchi S, Spicer AP, McDonald JA, Kimata K: Three isoforms of mammalian hyaluronan synthases have distinct enzymatic properties. J. Biol. Chem. 274, 25085-25092, 1999 |
| (6) |
Vigetti D, Karousou E, Viola M, Deleonibus S, De Luca G, Passi A: Hyaluronan: biosynthesis and signaling. Biochim. Biophys. Acta 1840, 2452-2459, 2014 |
2023年 6月15日
|
|---|








