この作品はクリエイティブ・コモンズ 表示 4.0 |
 |
|---|
グリコサミノグリカンの分解機構 | ||||||||||||||||||||||||||||||||||
 |
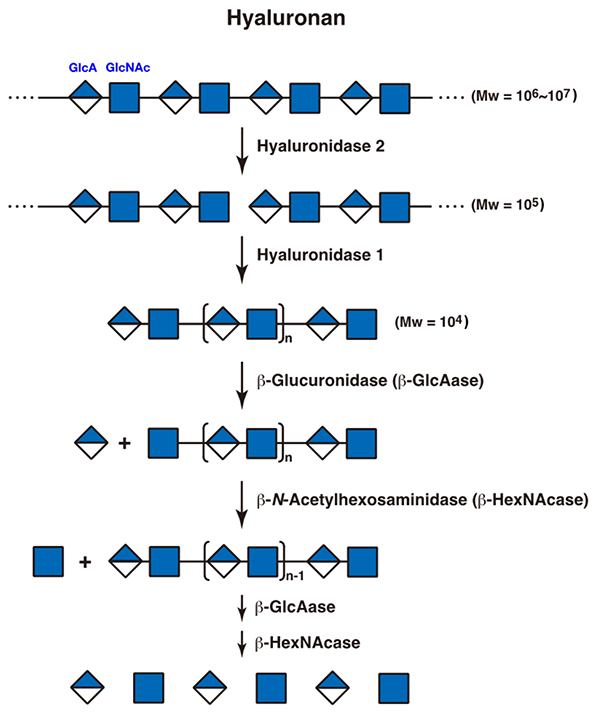
グリコサミノグリカンの代謝分解の主要な部分は、リソソームで起こる。エンドサイトーシスで細胞内に取り込まれたグリコサミノグリカン(あるいはプロテオグリカン)は、まずエンドソーム内あるいは初期リソソーム内でエンド型分解酵素によって加水分解を受ける。これは、高分子のグリコサミノグリカンをある程度低分子化することとプロテオグリカンの場合は、コアタンパク質から切り離す役割があると考えられる。ある程度低分子化されたグリコサミノグリカンは、リソソーム内でさらにエンド型加水分解酵素の作用を受けてオリゴ糖となり、その後エキソ型酵素群の協調的なはたらきによって非還元末端側から順々に加水分解される。これらの酵素の先天的欠損によってムコ多糖症が発症する(「ムコ多糖症」の項参照)。以下、各グリコサミノグリカンについて、その代謝分解のされ方を示す。 1.ヒアルロナンヒアルロナンはその分子量が106~107と非常に大きい。細胞内に取り込まれたのち、エンドソームでエンド型のβ-N-アセチルグルコサミニダーゼと考えられているヒアルロニダーゼ2によって分子量105程度にまで低分子化されたのち、リソソームでエンド型のβ-N-アセチルヘキソサミニダーゼであるヒアルロニダーゼ1による分解を受け、分子量104程度のオリゴ糖となる。その後、エキソ型の酵素である、β-グルクロニダーゼとβ-N-アセチルヘキソサミニダーゼによる加水分解を受け、非還元末端から単糖ごとに切り出されて完全に分解される(図 1)(1)。ヒアルロニダーゼ以外のエンド型ヒアルロナン加水分解酵素として、TMEM2とCEMIP/KIAA1199が同定されており、最近の研究から、ヒアルロナンの代謝においては、ヒアルロニダーゼ1、2よりも重要な役割を果たしていることが示唆されている(2, 3)。
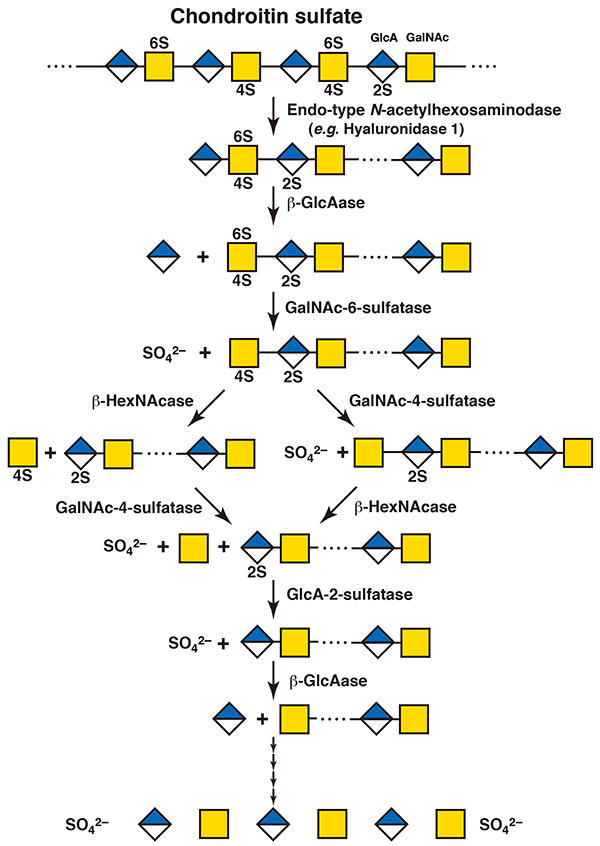
コンドロイチン硫酸もリソソームでヒアルロニダーゼ1による分解を受け、分子量104程度のオリゴ糖となる。これは、コンドロイチン硫酸とヒアルロナンの基本糖鎖骨格が似ているためである。その後、エキソ型の酵素群の協調的な加水分解を受け、非還元末端から単糖ごとに切り出されて分解されるが、どの順序ではたらくのかはオリゴ糖の構造による(4,5)。ヒアルロニダーゼ1による分解産物の非還元末端は、硫酸化されていないグルクロン酸であるため、まずエキソ型のβ-グルクロニダーゼが作用する。次に非還元末端となったN-アセチルガラクトサミン残基にエキソ型の酵素N-アセチルへキソサミニダーゼが作用するが、この糖残基が硫酸化されていた場合にはエキソ型のスルファターゼが先に作用する。4位が硫酸化されていた場合はN-アセチルガラクトサミン-4-スルファターゼが、6位が硫酸化されていた場合はN-アセチルガラクトサミン-6-スルファターゼが硫酸基を加水分解する。両方が硫酸化されていた場合には、まず先にN-アセチルガラクトサミン-6-スルファターゼが作用し、その後N-アセチルガラクトサミン-4-スルファターゼが残った硫酸基をはずす(5)。次に、エキソ型のβ-N-アセチルヘキソサミニダーゼがN-アセチルガラクトサミン残基を加水分解して遊離させる。一般にはグリコシダーゼがはたらく前にスルファターゼが硫酸基をはずすが、β-N-アセチルヘキソサミニダーゼはN-アセチルガラクトサミン残基の4位が硫酸化されていた場合でも作用しうることが知られている(5)。次からはこれらの繰り返しとなる(図 2)。グルクロン酸の2位が硫酸化されていた場合には、β-グルクロニダーゼが作用する前にグルクロン酸2-スルファターゼが硫酸基を加水分解する。ヒアルロニダーゼのうち、ヒアルロニダーゼ4はヒアルロナンには作用せず、コンドロイチン硫酸に特異的な加水分解酵素として同定されている(6)。コンドロイチン硫酸代謝におけるヒアルロニダーゼ4の役割はまだよく分かっていない。コンドロイチン硫酸に作用する細菌由来のエンド型スルファターゼの報告もあり(7)、これらはコンドロイチン硫酸の硫酸基を多糖の状態のまま加水分解することができる。ヒトにおいても同様の酵素が存在するのかは不明である。
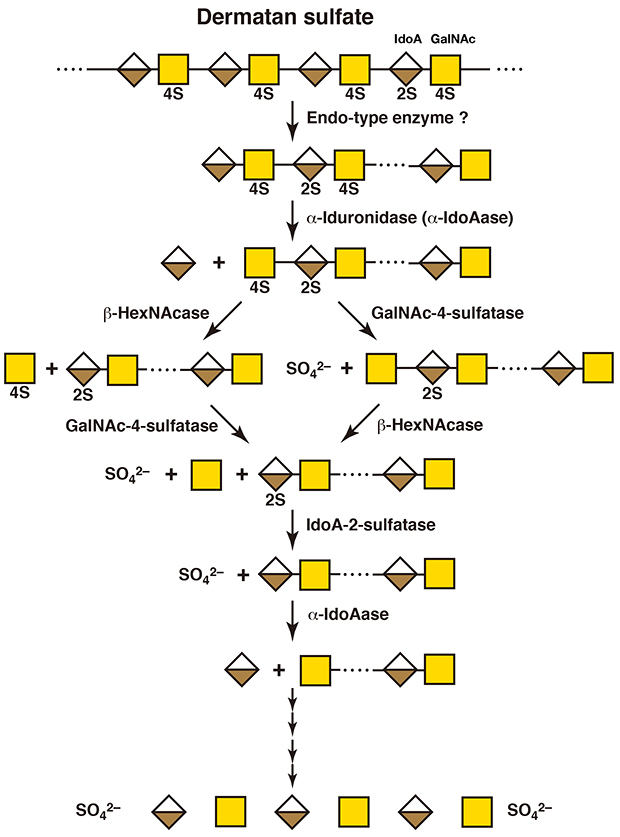
デルマタン硫酸に特異的に作用するエンド型の分解酵素はこれまでに見つかっていない。ヒアルロニダーゼはグルクロン酸の隣のN-アセチルヘキソサミニド結合のみを基質とするため、デルマタン硫酸には作用しない。デルマタン硫酸の代謝分解の際に、長鎖のままエキソ型の酵素が作用するのか、エンド型のイズロニダーゼやイズロン酸残基の隣であっても作用しうるエンド-β-N-アセチルガラクトサミニダーゼが存在するのかは不明である。ただし、デルマタン硫酸オリゴ糖の分解のされ方は、基本的にコンドロイチン硫酸の場合と同じである(図 3)(4,5)。イズロン酸が非還元末端にある場合には、エキソ型のα-イズロニダーゼが作用する。もしイズロン酸の2位が硫酸化されていた場合には、先んじてイズロン酸2-スルファターゼが作用する。
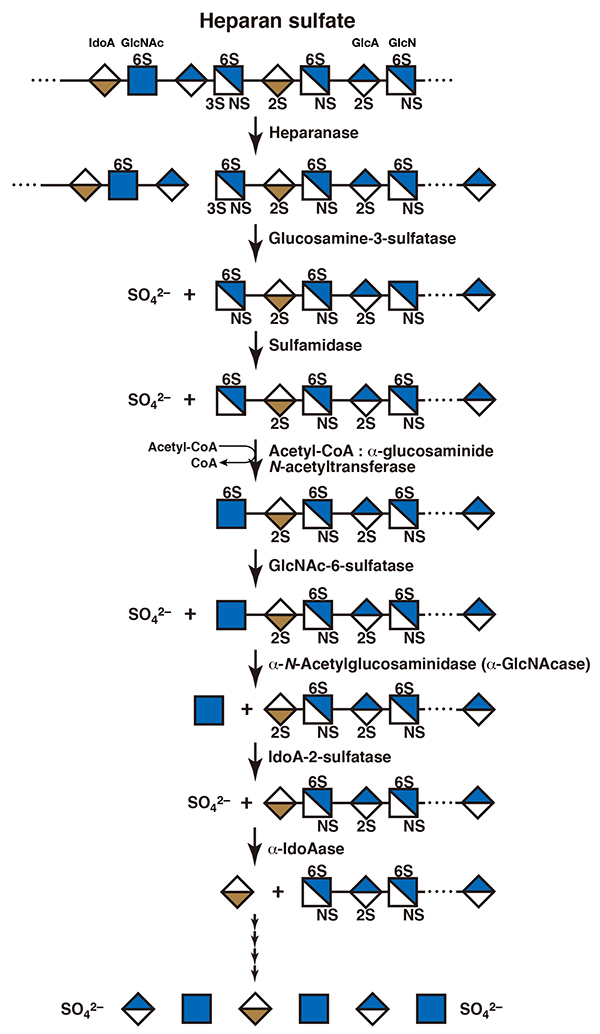
ヘパリン/ヘパラン硫酸の場合、まずエンドソームあるいは初期リソソームで、エンド-β-グルクロニダーゼであるヘパラナーゼが作用し、オリゴ糖にする。この後、リソソーム内でエキソ型の酵素群の協調的な加水分解を受け、非還元末端から単糖ごとに切り出されて分解されるが、どの順序ではたらくのかはオリゴ糖の構造による(図 4)(8)。ヘパラナーゼによる分解産物の非還元末端は、硫酸化グルコサミンであるため、まずエキソ型のスルファターゼが作用する。3位のヒドロキシ基が硫酸化されていた場合はグルコサミン-3-スルファターゼが硫酸基を加水分解する。ヘパラナーゼ作用部位のグルコサミンのアミノ基は硫酸化されているため、このあとスルファミダーゼが作用して、2位の硫酸基が加水分解される。これによって非還元末端糖残基はグルコサミンとなるが、このまま加水分解されることはない。グルコサミン残基はそのアミノ基の部分に、アセチルCoAからアセチルCoA:α-グルコサミニド-N-アセチルトランスフェラーゼによってアセチル基が転移され、一旦、N-アセチルグルコサミンとなる。このとき、もし、6位のヒドロキシ基が硫酸化されていた場合は、N-アセチルグルコサミン-6-スルファターゼが作用して、6位の硫酸基を加水分解する。ここでようやくエキソ型のα-N-アセチルグルコサミニダーゼが作用し、N-アセチルグルコサミン残基を遊離させる。続いて非還元末端となったウロン酸残基が硫酸化されていれば、イズロン酸2-スルファターゼあるいはグルクロン酸2-スルファターゼが作用し、その後、硫酸化されていないウロン酸をα-イズロニダーゼもしくはβ-グルクロニダーゼが加水分解する (8,9)。イズロン酸2-スルファターゼ、α-イズロニダーゼはデルマタン硫酸に作用する酵素と同じ酵素であり、グルクロン酸2-スルファターゼ、β-グルクロニダーゼもコンドロイチン硫酸やヒアルロナンに作用する酵素と同じである。一般に、エキソ型の酵素は基質特異性が低く、作用する単糖残基部分以外の構造はあまり認識しない。ヘパリン/ヘパラン硫酸の場合、多糖の状態で作用するエンドスルファターゼの存在が知られている(10)。これらは、へパラン硫酸6-O-エンドスルファターゼ-1(Sulf-1)、および-2(Sulf-2)と名付けられており、細胞表面において、ヘパラン硫酸プロテオグリカンの高硫酸化ドメイン中のN-硫酸化グルコサミン上の6位の硫酸基を加水分解する(「細胞外スルファターゼSULF1 およびSULF2」の項参照)。この硫酸基の除去によって糖鎖全体の立体構造が変化し、その結果、相互作用する生理活性タンパク質が変わり、ヘパラン硫酸の機能が調節される。
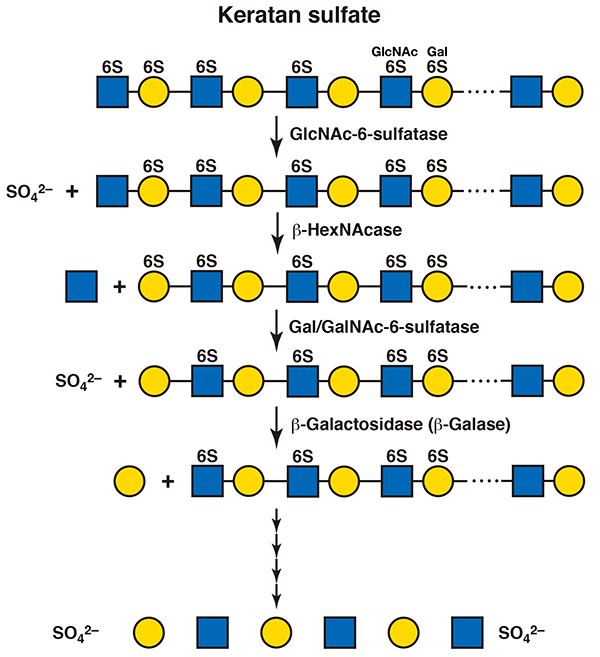
ケラタン硫酸の分解も同様に、エンド型加水分解酵素による低分子化からはじまると考えられる。エンド-β-ガラクトシダーゼの作用によってオリゴ糖が生じ、非還元末端のN-アセチルグルコサミン残基から順々に分解される。単糖単位を切り出すのは、エキソ型の酵素であるβ-N-アセチルヘキソサミニダーゼとβ-ガラクトシダーゼである(11)。非還元末端糖が硫酸化されていた場合には、まず、硫酸基が加水分解される。N-アセチルグルコサン-6-硫酸はヘパリン/へパラン硫酸に作用する酵素と同じN-アセチルグルコサン-6-スルファターゼにより(12)、ガラクトース-6-硫酸はコンドロイチン硫酸に作用する酵素と同じN-アセチルガラクトサミン-6-スルファターゼが作用する(13)(図 5)。
山田 修平(名城大学薬学部)
2023年 6月15日 | |||||||||||||||||||||||||||||||||