
氏名:細田 正恵
創価大学大学院 工学研究科で生命情報工学を専攻し、木下聖子教授のもとで2019年に学位取得後、創価大学の助教として教育と研究に従事しています。糖鎖構造のマルチプルアラインメントの研究をし、MCAWツールやMCAW-DBを開発しています。また、木下教授のGlyCosmosプロジェクトに参加しています。

氏名:安形 清彦
筑波大学大学院 生物科学研究科で学位取得後、La Jolla Cancer Research Foundation(現Sanford Burnham Prebys Medical Discovery Institute)にて、福田穰教授のもとで糖鎖生物学の研究について学ぶ。現所属(産総研)では、糖鎖遺伝子の発現解析、糖鎖の新しい機能の解析や糖鎖の応用について研究するとともに糖鎖DB(ACGG-DB)の開発に参画しています。
本シリーズの第4回では、レクチンに関するデータベース(DB)について説明いたします。一般的に糖鎖に結合する分子はレクチンと呼ばれています。植物に存在するレクチンは生物学においても長い間研究され、生理学的な活性の研究から細胞や糖鎖の研究用のツールとしても用いられています。動物の細胞や体内でも、糖鎖に結合する機能分子としてガレクチンやカルネキシンなどが重要な働きをしています。細菌から植物や動物まで様々な生物において、レクチンとしては知られていなくてもレクチン様活性を示す分子が数多くあります。ここでは、糖鎖研究に有用な様々なレクチンやその活性についてまとめているDB(LfDB, LM-GlycomeAtlas, MCAW-DB, GlyCosmos Lectins)を紹介します。
糖鎖の機能を理解する上で重要なテーマの一つは糖鎖と糖鎖に結合する分子(=レクチン)との相互作用です。糖鎖はタンパク質の修飾過程において品質管理に関わるレクチンのターゲットにもなりますし、細胞膜上では分子間や細胞間の結合の調節にも関わります。また動物の体内では、ホルモンなどの血中からのクリアランスや、リンパ球などの細胞移動やがん細胞の転移にも糖鎖と糖鎖結合分子が深く関わっており、糖鎖に結合する分子(=レクチン)の解析はますます重要になっています。また、菌類や植物の種子に含まれるレクチンは血液凝集性や毒素としても知られており、糖鎖結合活性を用いた研究用ツールとしても用いられてきました。 レクチンは様々な生物種に存在することやレクチン様活性と呼ばれる分子もあり、レクチンのデータベース(DB)はそれほど多くありません。レクチンの構造や配列を検索するためには、LectinDB 、UniLectinやGLYCO3D があります。また、レクチンもタンパク質ですのでUniProtKBや日本蛋白質構造データバンク などから情報を得ることも可能です。糖鎖との結合解析結果を含むDBは、機能解析を実施する上で有効です。なぜならレクチンは様々な糖鎖構造に結合し得る分子で、アフィニティの違いを理解して実験に用いる必要があるからです。糖鎖アレイを用いて測定した糖鎖結合活性の結果をまとめたMCAW-DB、糖鎖―レクチン結合をフロンタル•アフィニティクロマト分析法で測定したLfDB, そして最近開発されたマウス臓器のレクチンマイクロアレイ解析結果を示すLM-GlycomeAtlasなどがあります。
糖鎖に結合する活性は植物レクチンや動物レクチン以外にも報告され、レクチン様活性と呼ばれ、これらを含めたレクチンの数は年々増え続けています。例えば、immunoglobulin superfamilyに属する分子構造でシアル酸結合活性を有するSiglecや一部のポリペプチドGalNAc転移酵素(ppGalNAcT)が有するGalNAc-(O-Ser/Thr)結合活性などです。これらのように、レクチン活性に着目したデータベースがLectin Frontier Database(LfDB, Hirabayashi et al. 2015)で、日本糖鎖科学総合データベース(JCGGDB)の一つとして公開されました。現在では、表示方法などインターフェースや記載内容などを変更した最新版をACGG内で公開しています。ここではその特徴について紹介いたします。
※ LfDBはACGGからだけではなく、日本糖鎖科学コンソーシアム(JCGG)やGlyCosmos ポータルからも訪れることが出来ます。
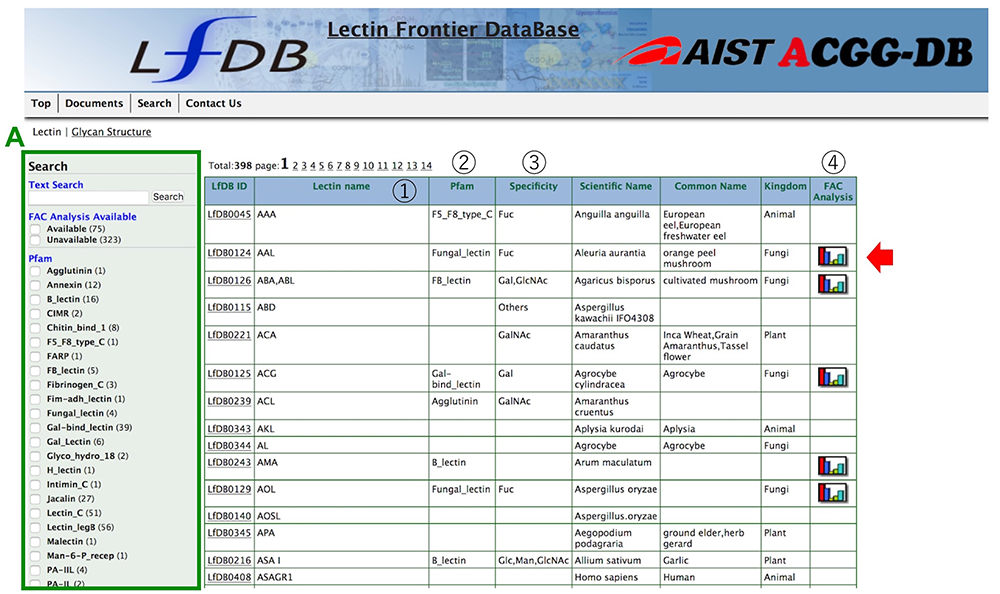
LfDBでは、現在398個のレクチンをリストしていますが、レクチンはアルファベット順に並べられ主に結合する糖鎖(単糖を含む)などの情報や検索ツールを表示しています(図 1)。糖鎖―レクチン結合をフロンタル•アフィニティクロマト分析法で測定した結果は75個のレクチンで見ることが可能です(Iwaki and Hirabayashi 2018)。検索モードとして、テキスト検索に加えファセット検索を用意してありますので絞り込み検索が簡単にできます。ファセット検索では、FAC Analysis Available, Pfam, Kingdom, Monosaccharide Specificityから選択することが可能です。
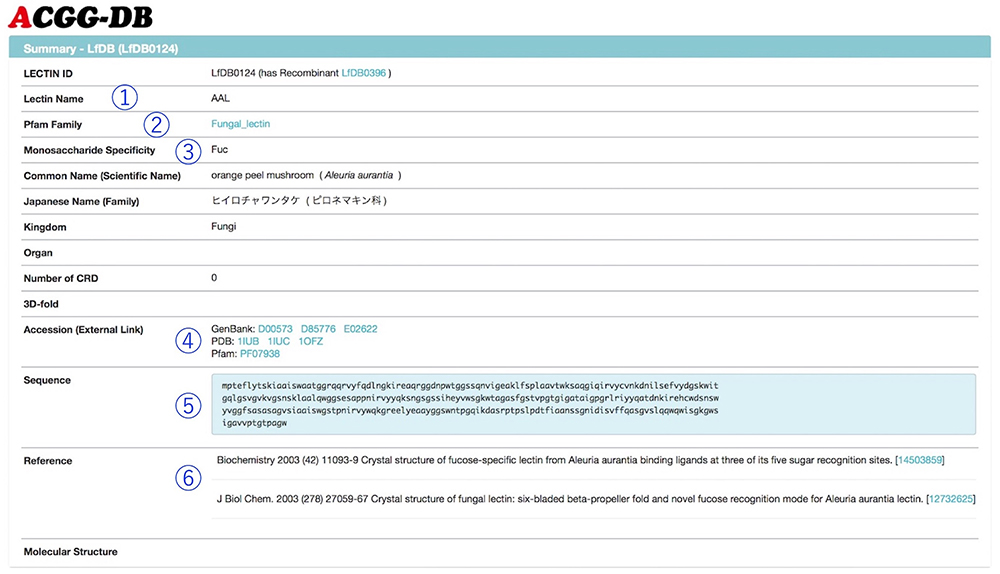
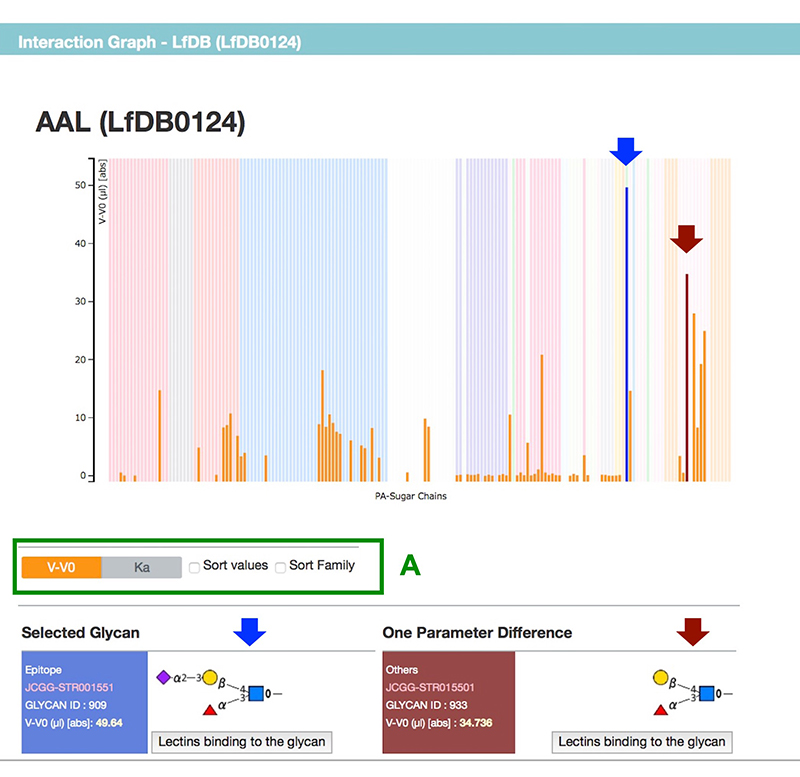
次に選択したレクチンのページを説明します。選択されたレクチンの各ページでは、サマリーに加えGenBankやPfamへのリンクと配列情報が示されます(図 2)。フロンタル•アフィニティクロマト分析法の結果はグラフで示され、各糖鎖に対するレクチンの結合能はV-V0あるいはKa値で表示されます(図 3)。各バーをマウスオーバーすることで、糖鎖構造と結合活性を見ることが出来ます。またバーをクリックして選択した場合、その糖鎖構造をグラフの下に表示するだけでなく、糖鎖構造の一箇所だけ異なる構造も表示します。これらの2糖鎖間で構造の違いとレクチン結合能の違いを比較することができ、レクチンを用いた実験の解釈に有用です。
レクチンをスポットしたアレイスライドを用いて解析するレクチンマイクロアレイ技術が開発され(Kuno et al. 2005)、微量なサンプルからの糖鎖構造の推測や、血清やがん細胞でサンプル間の糖鎖の違いを検出することが可能になりました。マウスの様々な臓器のスライドからレーザーマイクロダイセクション法を用いて抽出した糖タンパク質を、レクチンマイクロアレイ解析した結果をまとめたのがLM-GlycomeAtlasです(Nagai-Okatani et al. 2019)。このDBは正常なマウスの各臓器で発現している糖鎖構造を比較するのに有効で臓器特異的な糖鎖構造の傾向を知ることが出来ます。
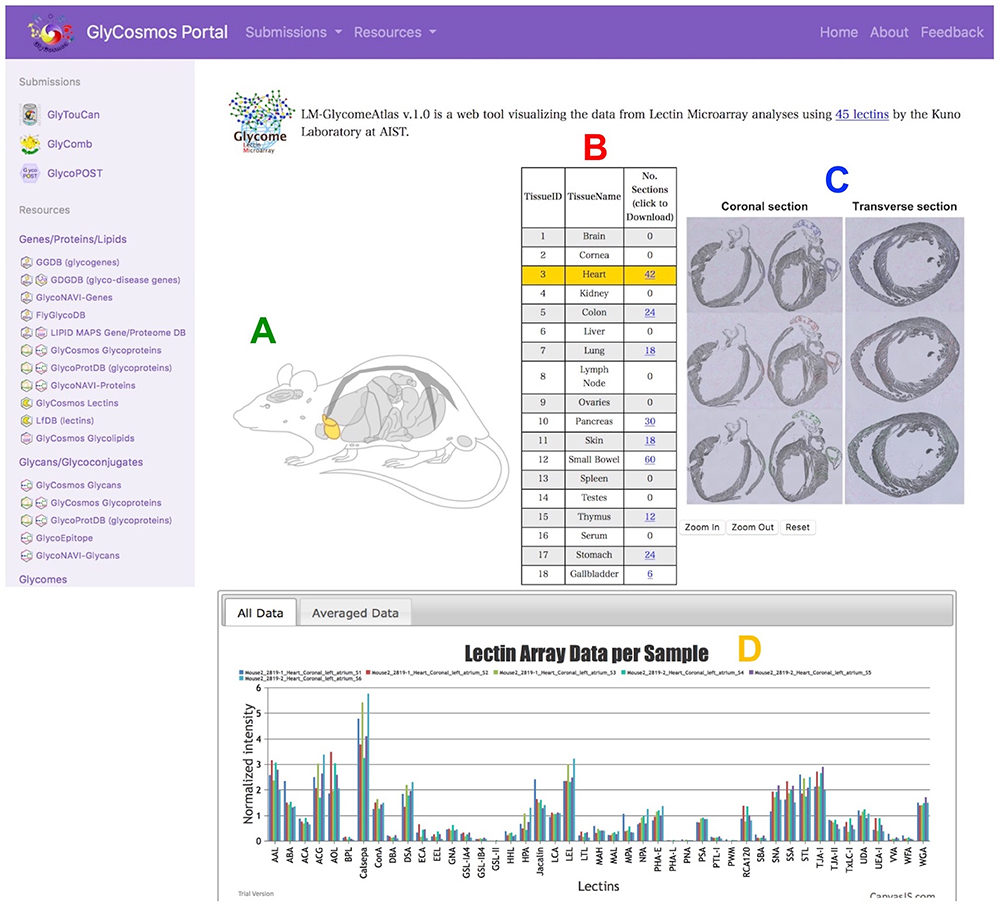
GlyCosmos Portal サイトからLM-GlycomeAtlasへと移動すると、マウスの組織図と18種類の臓器別の表が示されており、興味のある臓器(現在は9個)を選択することが出来ます(図 4)。臓器名(TissueName)を選択することで、臓器スライドの画像を表示し、サンプルのレーザーマイクロダイセクション後の画像を見ることも可能で、各サンプルの糖タンパク質の調製情報を得ることが出来ます。各マウスのレクチンマイクロアレイの結果もグラフで表示され、視覚的に結果を理解することが出来ます。測定結果などの詳細情報は表中のNo. Sectionsからダウンロードすることも可能です。
MCAW-DB (Hosoda et al. 2018) はhttps://mcawdb.glycoinfo.orgで利用できます。このデータベースはレクチンや抗体を含むGlycan Binding Proteins(GBPs)が認識する糖鎖について解析されたデータを格納しており、糖鎖アレイ実験結果を解析した計1081の糖鎖アラインメントデータが公開されています。糖鎖アラインメントはMultiple Carbohydrate Alignment with Weights(MCAW)tool(Hosoda et al. 2017) によりダイナミックプログラミング法を用いて、複数の糖鎖構造から共通部位を算出します。各糖鎖構造の同じ単糖や結合が揃うように比較計算を行なっているため、MCAW toolの解析結果では複数の糖鎖構造の共通部位を可視化できます。現在公開されているMCAW-DBは、Consortium for Functional Glycomics(CFG)で提供されている動物、植物、抗体、ウィルスや抗体などの糖鎖アレイ実験結果により高いRelative Fluorescence Units(RFU)値を示す糖鎖構造をMCAW toolで解析したデータです。このデータベースでは、GBPsが認識する糖鎖のパターンを閲覧することができます。
MCAW-DBのホーム画面は図 5のようにMCAW toolで解析した、CFGの糖鎖アレイ実験データのサンプル名や生物種などがリストとして表示されています。ユーザーがデータを検索するためのテキストサーチや、組織、タンパク質の分類などを絞り込める機能が画面左側にあります。
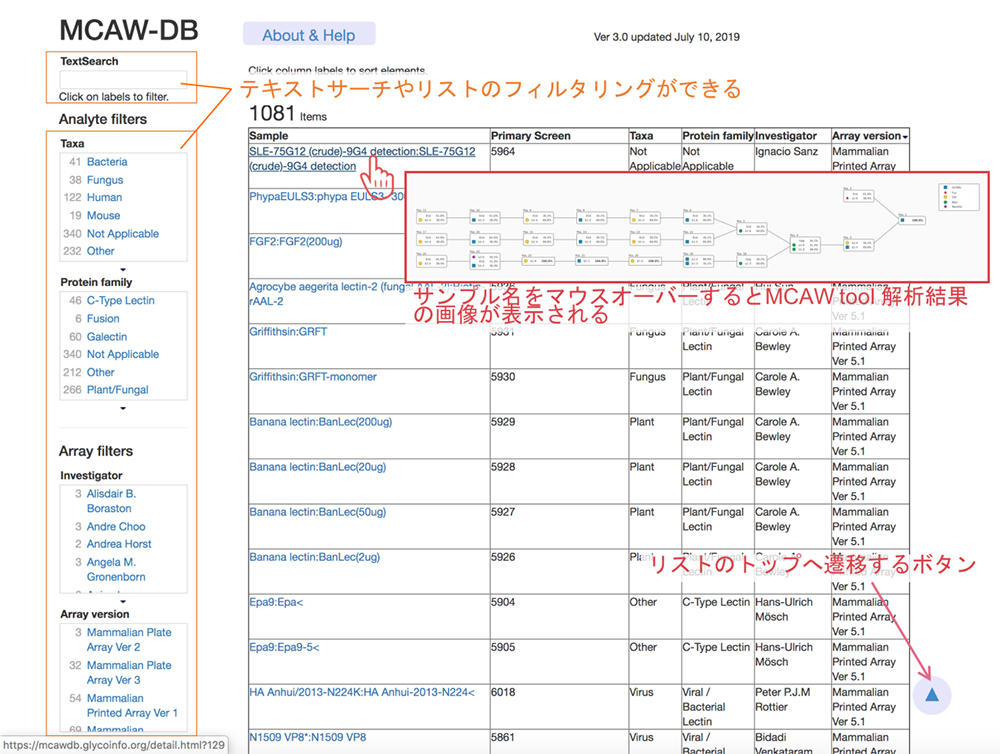
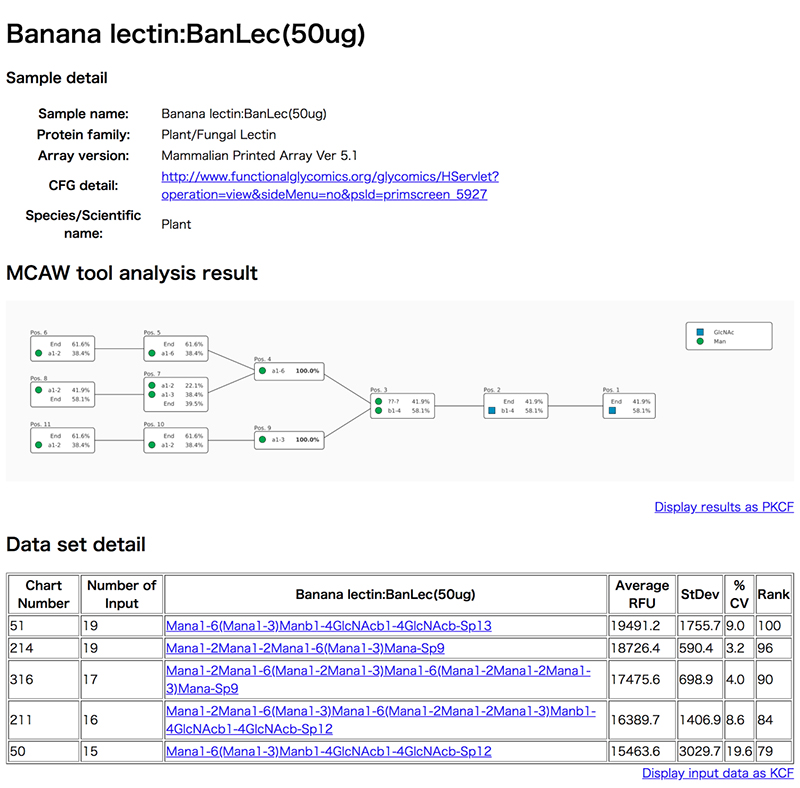
リスト中のサンプル名をクリックすると、詳細画面(図 6)が新しいウィンドウで開かれ、中央のMCAW tool analysis resultに糖鎖アラインメント画像が表示されます。詳細画面の上方にはサンプル名、タンパク質ファミリー、糖鎖アレイ実験のCFGのアレイバージョン、CFGの実験データの詳細リンクがあります。また、Data set detailではMCAW toolの解析に用いられたCFGの糖鎖アレイの糖鎖構造やRFU値などの情報が表示されます。糖鎖構造のリンク先にはGlyTouCanで登録されている構造が閲覧できます。MCAW toolの解析結果は、Data set detailにある糖鎖構造をアラインメントした結果です。共通する部位が割合で表現されています。100%と表示されている箇所は解析した糖鎖構造全てに共通して存在することを示します。図 6の場合、2分岐した高マンノース型N型糖鎖が共通する糖鎖構造であることがわかります。Endは糖鎖構造をアラインメントした際にアラインメントする単糖が無い場合に表示されます。
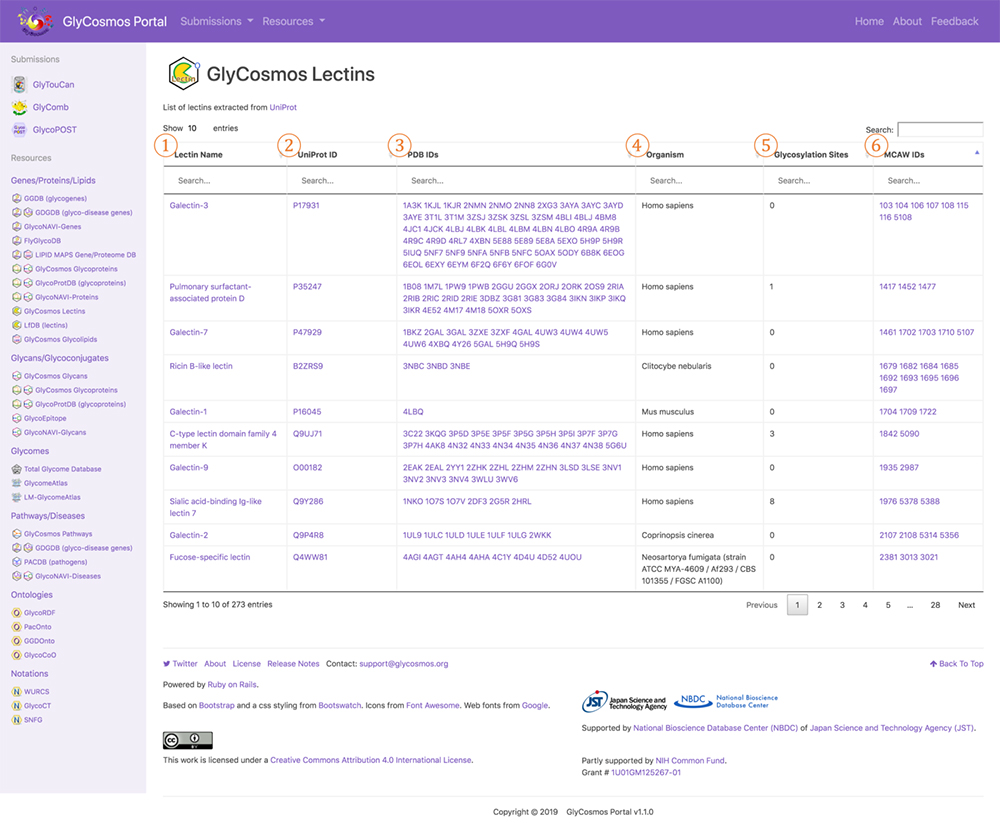
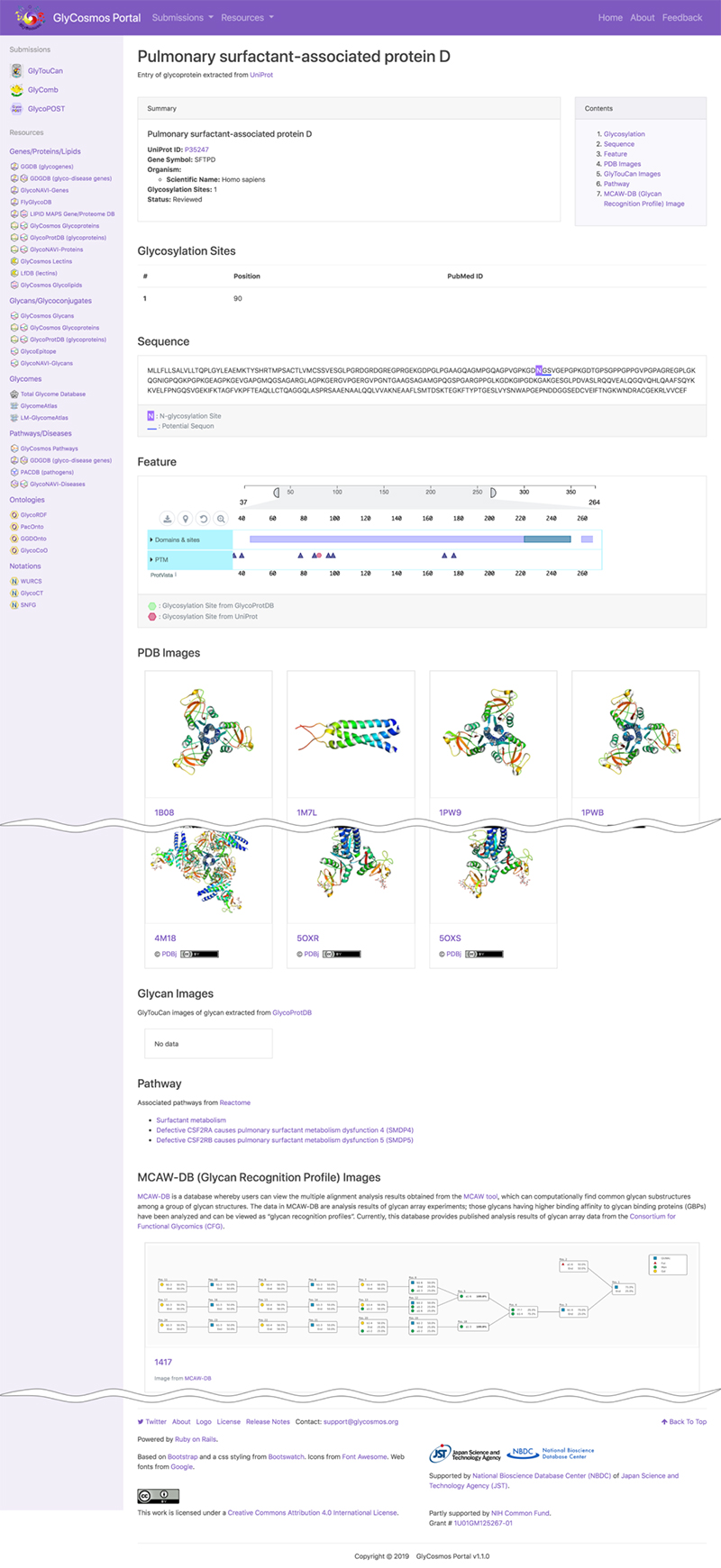
本シリーズGlycan and Databaseで紹介された GlyCosmos ポータルにて公開しているデータリソースGlyCosmos Lectinsは、UniProtにてレクチンと定義されているPDBのリストを提供しています。図 7のようにLectin name, UniProt ID, PDB IDsのカラムに並び、UniProtに登録されている Organism, Glycosylation Sites、さらに MCAW-DBのMCAW IDsをリストにしています。ここでは、セマンティックウェブ技術を用いてレクチンに関するデータの統合を可能にしています。レクチン名のリンク先には、レクチンのエントリーが表示されます(図 8)。UniProtに基づくレクチンの情報を閲覧できます。糖タンパク質の場合はその糖鎖付加部位、修飾糖鎖構造、PDBグラフィックが表示され、さらにレクチンのパスウェイ情報も閲覧できます。そしてGBPとしてMCAW-DBに登録されている場合はそのアラインメント画像が表示されます。
現在でも新しいレクチンが発見されたり改変レクチンが作製され、糖鎖研究にますますレクチンの有効利用が期待されています。本稿で紹介したMCAW-DBやLfDBにおいても新たなレクチンの活性が示されることが必要です。また、LM-GlycomeAtlasにおいても未解析の臓器のデータと合わせて糖タンパク質の解析も同時に進むことで、比較解析が可能となり糖鎖研究の進展に有用と考えられます。