この作品はクリエイティブ・コモンズ 表示 4.0 |
 |
|---|
シングルセル糖鎖・RNAシーケンス法 | ||||||||||||||
 |
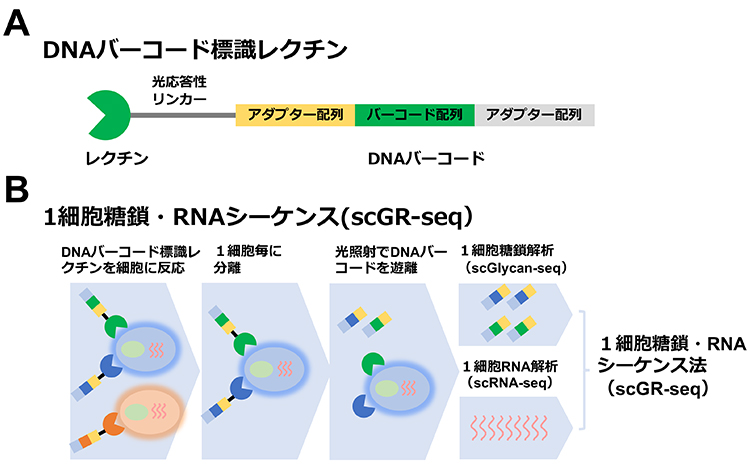
近年、1細胞ごとのオミクス情報を取得する技術が開発され、複雑な細胞集団の解析に変革をもたらした。1細胞ごとのトランスクリプトームを解析するためのscRNA-seqは生命科学研究に必須の技術として世界中で利用されている。糖鎖(グライコーム)は細胞の最表層を覆う生命に必須の情報分子であり、多細胞間相互作用を制御することで多様な生命現象に関与している。グライコームを解析する一般的な手法として、質量分析、液体クロマトグラフィー、キャピラリー電気泳動、レクチンマイクロアレイなどがある。しかしいずれの技術も細胞集団単位(バルク)での解析のみが可能であり、1細胞ごとの糖鎖発現を解析することができなかった。なぜ1細胞ごとに発現するグライコームを解析することが難しいのか?糖鎖情報は遺伝情報のようにポリメラーゼ連鎖反応(PCR)で簡単には増幅できないためである。そこで我々は複数のDNAバーコード標識レクチンを用いることで、糖鎖情報を遺伝情報に変換、PCRで増幅し、個々の細胞の糖鎖を次世代シーケンサー(NGS)で解析できるようになるのではないかと考えた(1)。
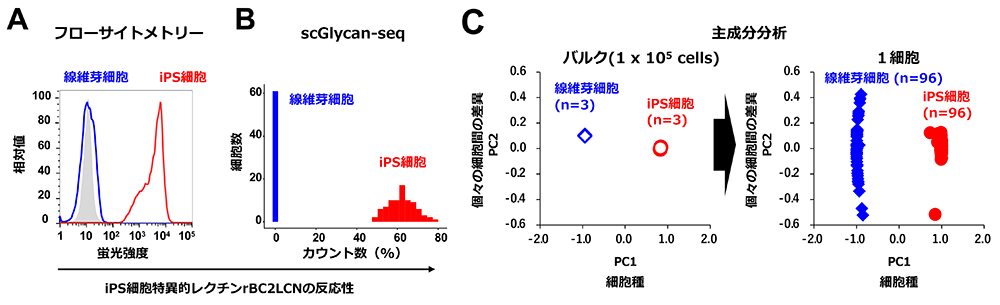
まずiPS細胞と線維芽細胞を用いてscGlycan-seq法の有効性を検証した。以前同定したiPS細胞特異的レクチン(rBC2LCN)は、フローサイトメトリー解析において、iPS細胞に結合し、高い蛍光強度を示した。一方、線維芽細胞には反応せず、蛍光は検出されなかった(図2A)。フローサイトメトリーの結果と同様にscGlycan-seq法においても、iPS細胞特異的レクチンはiPS細胞に対して高い反応性を示し、線維芽細胞には反応性を示さなかった(図2B)。そのためscGlycan-seq法を用いることで、標準的な方法と同じ結果になることがわかった。さらに主成分分析と呼ばれる統計解析手法を用いて、約40種のレクチンの反応パターンから細胞を分類した(図2C)。バルクデータにおいて、個々の細胞は細胞型ごと(iPS細胞と線維芽細胞)に明確に識別され、細胞型間の糖鎖発現の違いを検出できていることがわかった。しかし3つの反復試験試料(n=3)は全く同じところにプロットされており、個々の細胞間の差異は検出されない(図2C)。一方、シングルセルデータで細胞を分類したところ、細胞型間で明確に識別されただけでなく、個々の細胞間の差異も検出された(図2C)。さらにヒトiPS細胞から神経前駆細胞に分化する過程を解析すると、徐々に糖鎖プロファイルが変遷する様子が観察できた。ヒトiPS細胞特異的レクチンrBC2LCNの反応性は分化の過程で徐々に低下した。すなわちscGlycan-seq法を用いると、1細胞ごとの糖鎖発現の変化を定量的に解析できる。
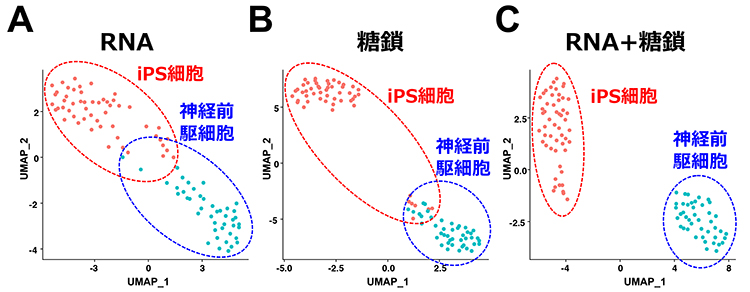
次にscGR-seq法を用いてヒトiPS細胞と神経前駆細胞を解析し、得られたデータをUMAP(Uniform Manifold Approximation and Projection for Dimension Reduction)と呼ばれる統計解析手法で解析した。いずれか一方の細胞情報(RNAもしくは糖鎖)では2種の細胞型(ヒトiPS細胞と神経前駆細胞)を完全には識別できなかった(図3A、B)。一方、糖鎖とRNA発現両方の細胞情報を用いることで、2つの細胞型を正確に識別できた(図3C)。また偽時間解析や部分的最小二乗回帰(PLS)により、ヒトiPS細胞特異的レクチンrBC2LCNが多能性遺伝子マーカー(POU5F1)と最も高い相関を示すことを確認できた。そして神経前駆細胞に対する新たな糖鎖マーカーとレクチンプローブを同定することができた。
従来のグライコーム解析技術では細胞集団に含まれる個々の細胞を予め分離して、少なくとも1万個以上の細胞を解析に用いる必要があった。また糖鎖遺伝子の発現は一般的に低く、scRNA-seqで1細胞ごとの糖鎖発現を解析することは難しかった。scGR-seqを用いると細胞集団に存在する1細胞ごとの糖鎖を一斉解析できる。そのため、これまでのグライコーム解析技術では難しかった細胞亜集団や希少細胞の糖鎖発現情報を取得できる。またグライコームとトランスクリプトームの関係を解明できることから、糖鎖の新たな機能解明が期待できる(3)。scGR-seqは細胞製品の規格試験・製造工程項目の設計や培養法の最適化、さらには新たな糖鎖創薬標的探索への応用が期待できる。糖鎖生物学において1細胞解析という新たなツールの誕生であり、シングルセルグライコミクス時代の幕開けと言える(4)。 舘野 浩章(国立研究開発法人産業技術総合研究所)
2023年 6月15日 | |||||||||||||