
氏名: Paul H. Weigel
Weigel博士は化学における理学士をCornell大学で、また1975年Johns Hopkins 大学医学部で博士号を得た。1978年助教授としてガルベストンのTexas大学医学部門に移り、1987年に生化学および細胞生物学の教授となった。1994年からはオクラホマシティにあるOklahoma大学健康科学センター医学部生化学および分子生物学科の教授と学科長を努めている。Weigel博士は幾つかの分野での重要な研究上の貢献をしているが、その中には受容体を介したエンドサイトーシスに対するmultiple coated pit pathwaysと、ヒアルロン酸生合成を担う酵素の発見が含まれる。博士は長年ヒアルロン酸の生化学と生物学に興味を持ってきた。彼のグループが初めてヒアルロン酸合成酵素遺伝子を同定し単離した。Weigel博士の研究室は、また特異的なヒアルロン酸レセプターと結合タンパク質を検出し研究するための高い比放射活性を有する、構造的に明確なヨウ素化ヒアルロン酸オリゴ糖を使って行う方法を開発した。このアプローチにより彼の研究室は最近、レセプターを介したエンドサイトーシスにより血中のヒアルロン酸と他のグルコサミノグリカンを除去するラットおよびヒト肝臓の内皮細胞のレセプターを精製、クローン化することに成功した。この膜レセプターにはHARE(Hyaluronan Receptor for Endocytosis)を含む幾つかの名前がある。
このシリーズの細菌のヒアルロン酸合成酵素に関する最初の総説(1998年)1では、ヒアルロン酸合成を担う酵素について、何が分かっているかについて論じた。1993年に我々がA群Streptococcus(連鎖球菌)からヒアルロン酸合成酵素をコードしている最初の遺伝子を発見した後、我々と多くの他の研究者は、他の細菌および広範囲な真核生物中で同様なヒアルロン酸合成酵素遺伝子を同定した2-6。これら20以上のヒアルロン酸合成酵素は1つを除いて共通な構造と作用特性を持ったタンパク質ファミリーを構成している。過去5年間、実際的に研究が進み、細菌のヒアルロン酸合成酵素の構造と機能、およびそれらの酵素がどのようにヒアルロン酸(以下HAと略す)を合成するのかについて理解が深まってきている。
現在我々は、種々の動物宿主に対する感染病原体の兵器工場の一部としてHA被膜を作る4つのタイプの細菌を知っている。HAの厚い層で自身を覆っている細菌は彼らの宿主の免疫システムから“隠れる”ことが出来る。これらの細菌としてはStreptococcus equisimilis菌(動物、時にはヒトの病原菌)、Streptococcus pyrogenes菌(ヒトの病原菌)、Streptococcus uberis菌(ウシの病原菌)とPasteurella multocida菌(鳥類の病原菌)がある。類似点および相違点から、既知のHA合成酵素は2つのカテゴリー、クラスI とクラスII に分類される6。Streptococcus菌類のHA合成酵素はクラスI ファミリーのメンバーであり、一方Pasteurella multocida菌のHA合成酵素(pmHAS)だけがクラスII ファミリーのメンバーである7。
クラスII pmHAS酵素は、構造、膜内トポロジーおよびHA産生の機構がクラスI HA合成酵素と大変異なる(表1)。クラスI とクラスII 合成酵素の間の主な構造上の違いとして、2種のタンパク質の膜ドメインの全体的な構成と分布の差がある(図1)。実験的に決定されたのは Streptococcus pyrogenes菌のHA合成酵素(spHAS)に関してのみではあるが8、クラスI ファミリーのメンバーは、始めから〜420アミノ酸の間に同一の局所的構成がある。クラスI 酵素は、アミノ末端近傍からカルボキシル末端近傍までタンパク質全体を通じて6-8箇所の膜貫通部分あるいは膜に結合したドメインを含んでいる。
表 1 クラスI とクラスII ヒアルロン酸合成酵素間の構造と酵素作用的な違い
| クラスI | クラスII | |
|
|
||
| ファミリーメンバー | 20以上のメンバーがクローン化(例 streptococcus、鳥類、両生類、ほ乳類) | 1つのみ(P. multocida) |
|
|
||
| サイズ(アミノ酸数) | 417-588 | 972 |
|
|
||
| タンパク質の膜への付着様式 | N末端からC末端にかけて6-8箇所の膜結合ドメイン有り | C末端で膜にアンカリング |
|
|
||
| 部分切断された可溶性タンパク質としての活性 | なし | 有り(例 1-703残基) |
|
|
||
| HA鎖の伸長部位 | 還元末端 | 非還元末端 |
|
|
||
| プライマーオリゴ糖を伸長する能力 | HAオリゴ糖が伸長した証拠はない | 容易にHAオリゴ糖を伸長する |
逆に、クラスII 酵素はカルボキシ末端のごく近傍に膜に未知のメカニズムにより接着したドメインを持っていて、たぶん別の膜結合分子との相互作用でタンパク質を繋ぎ止めている。その主たる機構の違いは2つのクラスの合成酵素では糖鎖の反対側の末端でHAを伸長することである。クラスII pmHASは非還元末端に新たな糖鎖を付加し、一方クラスI では還元末端側で糖鎖を伸長させる。クラスI 酵素と違い、クラスII pmHASは2つの糖転移酵素活性を持つ2ドメインモジュール構造を持ち、交互にHA鎖を結合、遊離して、新しい糖鎖を“古典的な”糖転移酵素メカニズムにより非還元末端に付加する。以下、本Updateの残り部分では、クラスI 合成酵素に関する新たな知見に焦点を絞ろう。
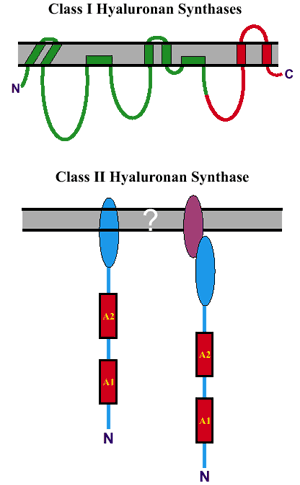
図 1 クラスI とクラスII ヒアルロン酸合成酵素間のトポロジーの違い
上図:streptococcal クラスI 合成酵素(緑色の線)は4箇所の膜貫通ドメインと2箇所の膜結合ドメインを含んでいる8。真核細胞のクラスI 合成酵素はこれより大きく2箇所の膜貫通ドメインの追加が予想される(赤色の線)。下図:クラスII HA合成酵素は膜に結合しているがそのメカニズムは未知である;nativeのpmHASのC末端で直接的に膜に結合しているのか(下の左側)、あるいはC末端ドメインで未同定の膜内分子と相互に結合しているのか(下の右側)、分かっていない。
A. 概説
HA合成酵素は細菌および真核細胞の膜結合型酵素で、活性型HA合成酵素は細胞内あるいは細胞質膜で見られる。最近の細胞内HAに関する知見9から活性型HA合成酵素は細胞の中にもあるらしいことが示されている。HA合成酵素は通常とは異なり、同一タンパク質内に2つの異なる糖転移酵素活性を持つ。さらにそれぞれの糖鎖が付加された後、新しく作られたHA鎖は次の糖鎖付加のための基質になる。歴史的には、一つの2糖単位の糖鎖がHA鎖へ付加する実質的な反応は、非還元末端で新しい糖鎖の付加が起きるためと言われてきた。事実は、これは2つの起こりうる反応メカニズムの1つでしかない。2つ目の反応機構 (図2A)はポリマーの還元端で2糖単位の糖鎖が付くことを表わしている。このメカニズムでは、伸長する末端でHA鎖にUDPが付着している。
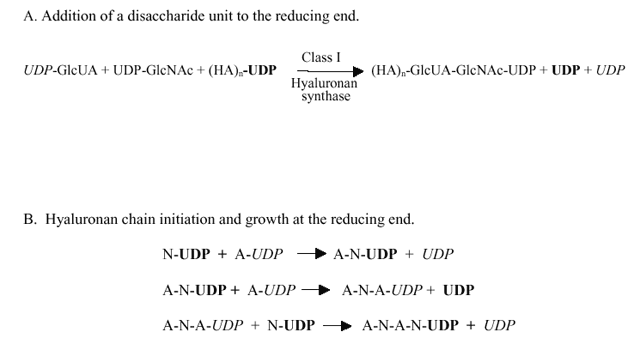
図 2 還元末端でのヒアルロン酸の合成機構
GlcUAとGlcNAcはそれぞれAとNで、UDP-糖鎖へのUDPグループはそれぞれイタリック体あるいは太字で示した。2糖単位の数はnで示した。ポリマー鎖へ付加されたUDP-糖鎖はそのまま、そのUDP結合が切断されることなしに転移される。それぞれの転移ステップでのUDP放出は前の糖鎖が付いた時形成されたHA-UDP中間体から生じる。このように正味2つのUDPグループは2糖単位が還元末端(A)で組み立てられた時遊離し、最後に付加された2つのUDP-糖鎖からは1つだけUDPが生じる。もう1つのUDPはHA-UDP鎖から生じる;新しい2糖単位が付着する前に最後に付加された糖鎖からである。 A;HAの還元末端での2つの糖鎖付加(2糖単位)。B; 還元末端でのHAの伸長開始および初期の伸長。
B. クラスI ヒアルロン酸合成酵素の多機能性
HHA生合成に必要な2つの活性化糖鎖前駆体はUDP-グルクロン酸 (UDP-GlcUA)とUDP-N-アセチルグルコサミン (UDP-GlcNAc) である。2糖ユニットを作り、HA鎖を伸長するために、クラスI HA合成酵素は少なくとも6つの異なる機能を有している(図3)。これらの活性には2つの異なる糖ヌクレオチドの結合部位、2つの糖転移酵素、伸長中のHA-UDP鎖に対する1つあるいは2つの結合部位、および酵素内部に糖鎖付加の次のサイクルを準備するためにHA鎖を移動する能力が含まれる。UDP-糖基質は細胞内で合成されHA合成酵素により使われ8、HA鎖は継続的に膜を越えて輸送されるので、産生されたHAは細胞の外側に押し出される。細菌の場合、このHAが被膜を作り、一方多くの真核細胞では細胞を取り囲む周辺細胞コートを作ったり、周囲の細胞外マトリックス中に放出したりする。
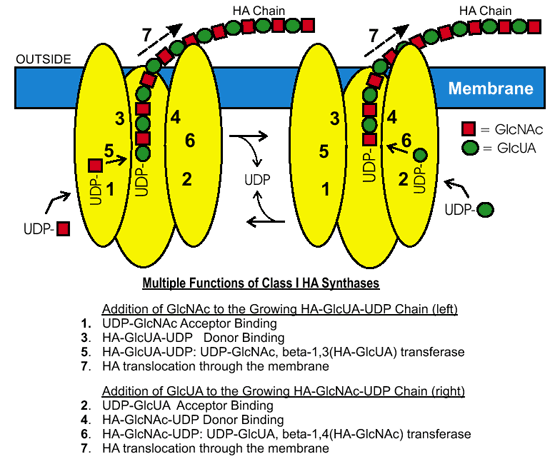
図 3 クラスI 酵素がヒアルロン酸の生合成を行うには多機能性が必要
図式では、膜結合型クラスI HA合成酵素と酵素が還元末端で2糖単位を付加してHA鎖を伸長させるのに必要なそれぞれの活性を示した。ヌクレオチド−糖基質が産生され細胞内の合成酵素によって利用されて、HA鎖は持続的に膜の外側に移動させられる。酵素が2つ描かれており、それぞれは酵素がどうやって交互にHAの2つのタイプのグリコシド結合を作るかを示している;同様の全ての基質が酵素に(一時に)結合し2糖が単位として協調的に付加されるというような交互糖鎖付加モデルも可能である。
C. クラスI ヒアルロン酸合成酵素は還元末端に付加させてHAを伸長する
(i) 背景 真核細胞からの膜分画を使って行った以前の2つの研究から10,11HA合成は還元末端で起こる事が示唆された。しかしながら細胞膜は他の糖転移酵素も含んでいるので、これらの結果は別の解釈が出来るかも知れない。また、これらの結果に対して3種のほ乳類HA合成酵素のアイソザイムがそれぞれ貢献する可能性は不明である。この重要な結論を確認するため、我々は2つの異なる精製した組み換えStreptococcus菌 HA合成酵素を使って同様の実験を行った。実験結果から、新たな糖鎖ユニットの付加は事実、還元末端で起こる事が証明された。我々は合成の方向を決定するために2つの実験を行った。ここでは予備的な形で記載されたもの12および他の論文で詳細に示されるであろう研究からの幾つかの実験証拠を要約して紹介する。
(ii) パルスラベル法によるアプローチ 最初の実験シリーズは、伸長するHA鎖が一ラウンドの糖鎖合成過程で早期あるいは後期にパルスラベルされるという良く確立された方法を用いた。これらの放射性ラベル化されたHA産物は2種のエクソ型糖分解酵素(すなわちβ-N-アセチルグルコサミニダーゼとβ-グルクロニダーゼ)で処理された。これらの酵素は非還元末端のみに作用し、個々のHA鎖から一度に1つの糖鎖を遊離する。精製したspHAS あるいはseHAS(Streptococcus equisimilis菌由来)のいずれかを用いた実験結果で、HA鎖に取り込まれる最初の糖鎖は最後に取り込まれる糖鎖よりも容易に遊離される、すなわちHAに取り込まれた最初の糖鎖が非還元末端に一番近いことが示された。
例えば、もしseHASによって産生されたHAがUDP-[3H]GlcNAcで素早く標識され、その後非標識の基質でインキュベートされた時、120分間の糖分解酵素処理で放射性標識の48%は遊離された(図4)。逆に標識化の順番を変えてHA鎖に付加される最後の糖鎖が標識されるようにした場合、放射活性のたった11%が遊離した。同様の結果が両方のUDP-糖鎖で、また両方のStreptococcus菌HASで得られた。最後に加わった糖鎖はポリマーの伸長終末に近いはずなので、これは還元末端となる。
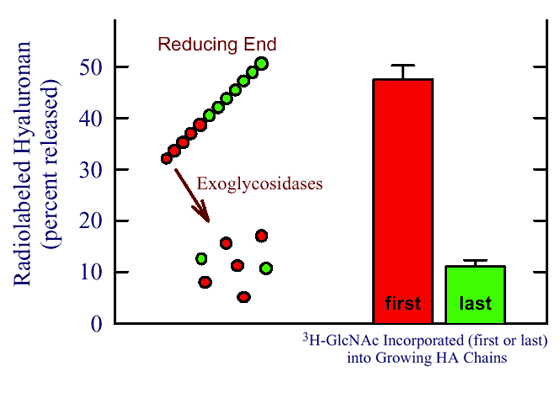
図 4 クラスI 酵素はヒアルロン酸の還元末端で糖鎖を付加する
HA鎖は、精製seHASによる1ラウンドの鎖長合成の早期(赤)あるいは後期(緑)のどちらかで、UDP-[3H]GlcNAcでパルス標識化した。放射性標識HAは非還元末端からのみ糖鎖を切り離すエキソ型グリコシダーゼで2時間処理し、遊離された放射活性を測定した。左側の図は早期あるいは後期に標識した場合のHA鎖中の標識GlcNAcの分布を表した図である。最初に付加された糖鎖は、もしそれらが還元末端で付加されたのならグリコシダーゼで容易に切断されることが予想される。伸長HA鎖が長くなるにつれ、最後の糖鎖は非還元末端から遠くなりグリコシダーゼで遊離され難くなる。
(iii) 動的ヒアルロン酸-UDP結合の証明 合成の方向を決めるための我々の第二の戦略は、UDPが伸長するHA鎖の還元末端に共有結合していることと、この結合が合成の進行に合わせて動的に切断されたり再形成されたりすることを示すことである。精製seHASとβ-32P標識UDP-GlcNAcを用い、HAに一定した再現性のある32Pの取り込みがあることを見出した(図5)。このような実験で回収される32P-HA量は、HA-GlcNAc-[32P]-P-ウリジン分子中の末端糖鎖のみが標識化されることから、例えばUDP-[3H]GlcNAcを用いた場合にHA鎖中の全糖鎖の50%が標識されるのに比べて比較的低い。
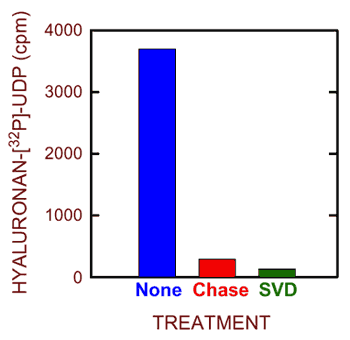
図 5 精製seHASによるヒアルロン酸—UDPの合成
精製seHASは非標識UDP-GlcUAと[32P]UDP-GlcNAcの存在下で25℃、1.5分間インキュベートした。サンプルはその後無処置(青)、過剰量の非標識UDP-糖鎖を加えて取り込みを停止させ“チェイス”(赤)、あるいは蛇毒phosphodiesterase(緑)で処理し、HAへの32Pの取り込みをペパークロマトグラフィーで定量した。酵素なしのバックグラウンドは差し引いた。
にもかかわらず、32Pで標識されたHAは容易に検出された。この32P標識HAが還元末端に共有結合しているUDPの存在を反映しているという結論は、色々な対照を用いることで支持される。例えば、標識されたものはヒアルロニダーゼや蛇毒phosphodiesteraseで処理すると破壊された(図5)。最も重要なことは放射性標識が、非標識UDP-糖鎖の添加により除去されたことである。この“チェイス”対照から伸長は還元端で起こることが確認された。なぜならこの末端での合成に対するメカニズムでは(図2B)、次の付加された糖鎖は、結合しているUDPがそれ以前に付加された糖鎖のものと置換することを要求するからである。このように[32P]UDPグループは、次の糖鎖が付加されてHA-GlcNAc-GlcUA-P-ウリジンに加わった時、HA-GlcNAc-[32P]-P-ウリジン鎖から失われる。無処置の場合は、次の糖鎖添加で再び放射性標識産物(すなわちHA-GlcNAc-GlcUA- GlcNAc-[32P]-P-ウリジン)が形成されるが、過剰の非標識UDP-GlcNAc存在下では32Pのこの後者産物への取り込みは著しく低下する(図5)。
D. 還元末端でのヒアルロン酸糖鎖の伸長と命名法
還元末端でのHA糖鎖が伸長している間、UDP-糖鎖は供与体ではなく(非還元末端で起こる付加の場合は供与体)、むしろ受容体である。従って、UDP-GlcNAcを利用するクラスI HA合成酵素転移活性は、事実GlaUA(β1,3)GlcNAc結合を形成する。逆にクラスII pmHAS活性はUDP-GlcNAcを利用して別のGlcNAc(β1,4)GlcUA結合を形成する。クラスI HA合成酵素の多機能性は、酵素が還元末端でどのようにHAを結合させるかの推論から図3に要約した。還元末端で1つの糖鎖が付加される各サイクル中で放出されるUDPは以前に付加された単糖鎖からのものである(図2B)。転移される供与体の種類はHA鎖であり、やはりUDPに結合することで活性化されている。
このような転移活性に対する系統名は、IUBMB命名法に従って、供与体:受容体、転移される基と規定されるであろう。従ってGlcUA残基を伸長するHA鎖の還元末端の GlcNAcに結合させる活性は:
(HA)-GlcNAc-UDP: UDP-GlcUA, β(1,4)-hyaluronyl transferase
同様にGlcNAc-UDPを(HA)-GlcUA-UDP鎖へ結合させる活性は:
(HA)-GlcUA-UDP: UDP-GlcNAc, β(1,3)-hyaluronyl transferase となる。
E. 還元末端でのヒアルロン酸合成の生物学的意味
還元末端で鎖長の伸長が起こる場合、HA産物は最後の糖鎖に付いたUDPを持たねばならない。HA-UDP結合の加水分解が起こると、HA鎖はもはやUDPとの結合による活性化は起こらず転移酵素反応の供与体として供されることが出来なくなるので、鎖長伸長は止まる。反対側で起こる非還元末端での鎖長伸長に対して、還元末端のoriginal-UDP連鎖は鎖長伸長に関与しない。もしこの結合が加水分解されても、ポリマーの反対側末端での鎖長伸長には影響しない。
2つのHA-UDP結合は、HA中の2つのグリコシド結合よりも、生理的条件下で不安定であるので、中性領域であっても加水分解に対して感受性がある。従って種々の方法で調製された市販品のHA鎖は、それらの還元末端にもはやUDPを含んでいることはないだろう。しかしながら、新たに放出されたHA鎖の還元末端の新規なHA-UDP構造要素の存在が、この結合を認知する様にデザインされたタンパク質に対して特異的な認識部位を提供出来るのだろうと仮定してみることは興味深い。このような相互作用の意味は生物学的には重要であろうが、まだ調べられていない。
Ellis Kempner博士との共同研究で、我々は活性体Streptococcus菌HA合成酵素が単体なのかもっと大きいオリゴ体なのか決定するために放射線不活性化分析を用いた14。Native spHASあるいはseHASを発現しているStreptococcus菌細胞から調製した膜と、各組み換え酵素を発現している大腸菌から調製した膜を分析した。これら4つの実験結果は、両Streptococcus菌からの活性のあるnativeあるいは組み換えHA合成酵素は1つのHA合成酵素タンパク質を含んでいたが、HASタンパク質は分子量約23kDaの追加部分があった。同様の結果が後にXonopus HAS酵素でも得られが、この場合追加分子量については決定されなかった15。幸運にも我々の研究では、カルジオリピン(ジホスファチジルグリセロール)としてこの追加分子量を同定することが出来た。従って膜の活性体クラスI HA合成酵素は、他の構成成分と複合体になっている単量体:seHASあるいはspHASでは、この活性体複合体〜16カルジオリピン分子と結合したHASタンパク質である。
HA合成酵素そのものが膜を通り越すHAの転移輸送に関与している図3のモデルは、種々の生化学的および遺伝子学的データを基にしている1-3。これらの研究はin vitroでStreptococcus菌HA合成酵素がHA合成に必要な唯一のタンパク質であることを示した。また、合成酵素をコードしているhasA遺伝子は、通常はHAを作らないBacillus subtilisやEnterococcus faecalisの様な細菌類にHAを作るように形質転換させるのに必要な唯一の遺伝子である。もしUDP-糖鎖の前駆体がこのような細胞中にあれば、hasA遺伝子で形質転換させた細菌は細胞質の前駆体を使ってHAを作り、それをHA被膜や培地中に蓄積する分泌産物として細胞外に送り出すことができるようなる。これらと他のデータを基に考えると、多糖体のトランスポーターがHAを細胞の外側に出現させるために必要であると示すものはない。
A.概説
元の総説の第5章が掲載された時、細菌あるいは真核生物のHA合成酵素を界面活性剤で可溶化し、精製に成功した報告はなかった。どんな材料からのHA合成酵素も、活性を保ったまま界面活性剤で可溶化し精製することは非常に困難であった。しかしながらその後、1つの真核細胞と3つの細菌性HA合成酵素が精製された5-7,16,17。HAが発見されて64年後、遂にA群とC群 Streptococcus菌のHA合成酵素が大腸菌で発現されたことで、これらの酵素を精製することが出来た。組み換えStreptococcus菌の酵素は最も容易に精製されたクラスI HA合成酵素であるので、最も広く研究されている。クラスII P. multocida 菌HA合成酵素は、大腸菌発現組み換え体の、一部が切断された可溶型のタンパク質として精製された6,7。遂に、組み換えマウスHAS1酵素が精製され、動態学的に特性が明らかになった5,17。他の真核生物のHA合成酵素は精製されていない。Streptococcus菌 HA合成酵素は、夾雑物なしの状態で研究出来るヘテロな多糖体ポリマーを作る最初の酵素である。
B. ヒアルロン酸合成酵素はヒアルロン酸の生合成に必要な唯一のタンパク質である
ヒヒアルロン酸の生合成はたった1つの遺伝子産物だけが必要であるとの発見は、HA合成過程が多機能性であり(図3)、かつ複雑であったので当初は驚きであった。例えば多くの研究者は、少なくとも2つの酵素が2つの糖転移酵素の反応を触媒するために関与しているだろうと予想していた。我々は、合成酵素は図3中に示された6-7の機能を有するためには〜49 Kdaより大きいと予想した。にもかかわらず遺伝学的、生化学的および放射線不活性化の研究、そして精製した酵素を用いた研究の結果を基にすると、UDP-糖鎖を供給された時 —HA合成酵素がHAの生合成に必要な唯一のタンパク質である、ことが明らかである。HA合成酵素がHAを生合成するためにプライマーや他のタンパク質を必要とする、との証拠はない。
C.ヒアルロン酸合成酵素タンパク質の特性
3つのStreptococcus菌HA合成酵素タンパク質のアミノ酸配列は〜70%の同一性がある。真核生物HA合成酵素間のアミノ酸配列の同一性は〜50%から95%の範囲である。Streptococcus菌タンパク質は約35%大きい真核生物タンパク質に対し、〜25%の同一性がある。PmHAS酵素に対するものを除き、全てのHA合成酵素タンパク質は今日まで共通の多くのアミノ酸モチーフを持っていると特徴づけられ、そして多くの予想される膜貫通型ドメインを持つ膜タンパク質である1-3。HA合成酵素タンパク質は非常に小さく緊密に折りたたまれているらしい。強力な界面活性剤であるドデシル硫酸ナトリウム存在下の電気泳動中でさえ、HASタンパク質は本当の大きさよりも〜15%小さい様な挙動をする。還元剤でこの挙動が変わらないから、ジスルフィド結合はたぶん無いことが示される。ジスルフィド結合がないことは、システイン残基を4あるいは6個のしか持たないStreptococcus菌酵素で生化学的に確認された18,19。また、後者の研究でシステイン残基はHA合成酵素活性には不要であることが示された。システインを欠損したA群とC群 Streptococcus菌 HA合成酵素では、野生型酵素活性の〜20%から60%が残っている。同様の研究はいずれの真核生物HA合成酵素ではまだ行われていない。と言うのは、これらのより分子サイズの大きな酵素ではシステイン残基数が3倍もあり、同様のアプローチをすることは技術的にさらに難しいことが理由の一つである。
D. ヒアルロン酸合成酵素の酵素動態特性 違いはあるが、色々な生物の膜にあるHA合成酵素の酵素学的動態は一般的に非常に似ている。UDP-GlcUA利用に対するKm値は例えば Streptococcus菌とヒトHA合成酵素に対しては30-75 μMで、概してUDP-GlcNAc利用に対してのそれ(例えば spHASと3つのヒト酵素に対して150-1000μM)よりも低い。HA合成酵素でUDP-GlcNAcの方の利用効率が悪いのは、この前駆体糖鎖がUDP-GlcUAよりも、多くの、より重要な構造の生合成に使われる事実を反映している。例えば細菌での細胞壁の生合成あるいは真核生物の糖タンパク質のN-結合型オリゴ糖におけるコアキトビオシル結合である。もし、UDP-GlcNAcへのKm値の方が低ければ、HA合成がこの前駆体を使い果し細胞性の複合糖質生合成が下がってしまうだろう。我々はこの考えに矛盾しないspHAS 活性対UDP-GlcNAc濃度のS字型Kmプロファイルを認めた20。この予期せぬ発見はUDP-GlcNAcがHA合成酵素のアロステリック制御因子かもしれないということを示している。SeHASや真核生物HA合成酵素の類似した挙動に対するin vitroでの酵素動態的証拠はないが、in vivoでの挙動は、特に多数の小分子によるアロステリックな制御については異なる可能性がある。
研究者それぞれが別の方法を使ってHA合成量をHASタンパク質量に基準化するので、種々のHA合成酵素に対するVmaxを比較することはさらに困難である。分離された膜中のStreptococcus菌HA合成酵素のHA重合率は〜1200-2400糖鎖/分と計算された。この伸長率だと、活性HA合成酵素1分子(あるいは脂質との複合体)は2 MDaサイズの1本鎖HAを作るのに8-16分を要する。生きた細胞中のHA伸長率は決定されていないが、in vitroで測定されたものより速いと思われる。
外因性のリン脂質なしで単一型高分子の状態として精製した場合、Streptococcus菌のヒアルロン酸合成酵素の活性は非常に低い16。しかしながら、カルジオリピンを再添加すると、酵素活性は10倍にも上がる(図6)。 SpHAS酵素はカルジオリピンで特異的に活性化され、一方seHASはホスファチジルセリンでも活性化される。これらの結果に基づき、他の研究者は界面活性剤で可溶化したクラスI の真核生物HA合成酵素の活性を維持するのに種々のリン脂質を使って試みた。残念なことにこれらはまだうまく行っていない。これら種々の真核生物HA合成酵素、特に3種のイソ型ヒトHA合成酵素精製の失敗はこれまでこの分野の失望であり、限界である。
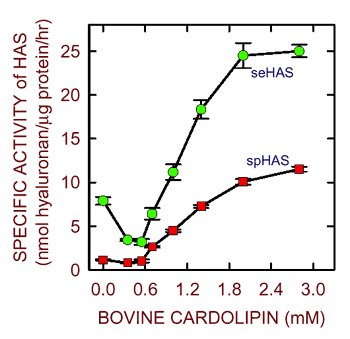
図 6 カルジオリピンは精製Streptococcus菌HA合成酵素の活性を刺激する
精製組み換えseHAS(緑)とspHAS(赤)酵素16の活性は外因性の脂質の非存在下では低かったが、ウシ由来のカルジオリピン混合物の添加量を増やすことで刺激された。
にも関わらず、我々は最近別の脂肪酸を含む合成カルジオリピンは精製seHASに対する活性化能が全く異なることを発見した(図7)。例えばC14のアシル鎖脂肪酸(ミリスチン酸)だけを含むカルジオリピンはseHASの活性化が全く出来ないが、C18(Δ9) アシル鎖脂肪酸(オレイン酸=炭素9,10に二重結合を持つパルミチン酸)だけを含むカルジオリピンは、大腸菌あるいはウシ肝臓から調製された市販のカルジオリピンで通常観察されるよりも大幅に活性化した。このようにクラスI HA合成酵素ファミリー内で、活性化に必要なリン脂質は特に差があるらしいが、これら全てのHA合成酵素はHA合成のために1,2個の特殊なタイプのリン脂質を必要とする可能がある。我々は、リン脂質分子は単量体タンパク質が穴の様な通路を形成しそこを通じて伸びつつあるHA鎖が通過して行くことを助けると言う概念を提言する16。例えばカルジオリピンの脂質部分は脂質の二重層やHA鎖の疎水パッチと相互作用するが、一方でカルジオリピン分子の酸性の頭部基は酵素やHAの親水性部分と相互作用する。
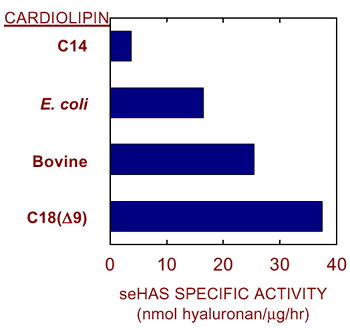
図 7 カルジオリピンのタイプに依存したStreptococcus菌ヒアルロン酸合成酵素の活性化
精製組み換えseHASは、合成調製物(Avanti製C14あるいはC18(Δ9)脂肪酸)、あるいはウシ肝臓あるいは大腸菌の膜から抽出した2 mMカルジオリピン存在下で、異なる活性を示す。
1998年当時、HA合成酵素の機能についての主要な未解決問題の一つは、リン脂質複合体であるたった一つのタンパク質がHAを形成出来、また新しく合成した多糖体を細胞膜を通して細胞外へ送り出すことができるという発見をどう矛盾なく説明出来るかということであった。我々は酵素がどのようにしてHAの合成と移動を成し得るかを説明する振り子仮説を作り上げた。幾つかのバリエーションがある振り子仮説の詳細は別のところで述べるが、その考えを動画(ここをクリック)で伝える。最初の方では、伸びつつあるHA鎖が見て分かるように合成酵素の前に示されている。後の方では最後の数個の糖鎖付加が繰り返され、HA合成酵素タンパク質−脂質複合体中の穴から通り抜けるHAが見える。
クラスI HA合成酵素がどのように働くかが説明可能な振り子仮説の鍵となる特徴は以下の通りである。
1998年では未解決であったStreptococcus菌HA合成酵素に関する幾つかの疑問については、即ちこれらの酵素が還元末端からHAを作ること、そしてHA合成が始まるのにプライマーは不要であることは、その後回答がなされた。HA合成中に共有結合の酵素中間生成物が産生されるかについてはまだ証拠がない。研究の進展にも関わらず、クラスI 合成酵素がどの様に機能するかについて少なくとも3つの基礎的問題が未解決のまま残っている。
A. HASタンパク質中に幾つのUDP-糖鎖結合部位があるのか?
主要な残された問題の1つは、タンパク質中に2つの別のUDP-糖鎖結合部位があるのか、あるいは部位は1つで交互に特異性が変わるのかである。この後者モデルは変わったものであるが、一考に値する。クラスI 合成酵素は一つだけ典的“DXD”モチーフ(大部分の糖転移酵素のヌクレオチド−糖鎖結合部位の重要な部分である)を持つ。もしこれらの酵素中の関連領域が2つ目のヌクレオチド−糖鎖結合部位を提供しないなら、一部位モデルの可能性が高くなる。この場合、1つの共通結合部位がその糖鎖結合特異性を変化させ、2つの基質が交互に認識されると考えられる。このような新規のメカニズムでは相当大きな立体構造の変化と、たぶん酵素内部でのかなり大きな動きが必要とされるだろう。
B. HASタンパク質中に幾つの糖転移酵素活性部位があるのか?
関連した残された問題は2つの糖転移酵素活性部位があるのか(例クラスII 酵素のように酵素の別々のドメインに)、あるいは万能な部位が1つだけあるのかで、その場合受容体と供与体に対する特異性が交互に変わり方法で2つの糖鎖結合が作られる。
C. どのようにしてヒアルロン酸産物の大きさは制御されるのか?
我々は今、HAのサイズは細胞とその生物活性との相互作用に影響を及ぼすらしいことを認識している。異なるHA合成酵素が異なる分子量分布のHAをどうやって、またなぜ作るのか、非常に興味深いにも関わらず、HA鎖長を制御する酵素の性質についてはほとんど知らない。我々と他の研究室は、特殊なHA合成酵素の突然変異が変異体を創り出し、そのものは違うサイズのHAを作ることを証明したが、鎖長の制御に関しては分かっていない。
明らかに、もし我々がどのようにしてクラスI Streptococcus菌や真核生物HA合成酵素がHAポリマーを合成するのかについて分子レベルでの詳細を理解したいと思うなら、これらの問題および他の残された質問に答えることは非常に重要である。願わくば、これらの質問の幾つかが解決され、将来再びupdate版の本章で論じたい。
私は、どのようにしてHA合成が起こるかについて、分子レベルで幾つかの基本的で重要な質問への回答が得られ始めた過去5年間の研究成果を述べた。1993年、我々はグリコサミノグリカン合成酵素での初めてのクローニングを報告した。2004年現在20以上のHA合成酵素が同定、クローン化され、多くは組み換えタンパク質として発現されている。多くのクラスI ファミリーの最小メンバーであるStreptococcus菌HA合成酵素の研究が、分子レベルでのHA生合成の詳細を理解するための最良の機会を提供し続けている。
謝辞
この総説に書かれた著者の研究はthe Institute of General SciencesからのNIH補助金GM35978で支援された。