氏名:山田 一作
1997年に東京都立大大学院工学研究科にて博士号取得。名古屋大学(日本学術振興会特別研究員(COE))などを経て、2002年から野口研究所研究員、2006年から同所にて糖鎖科学研究を開始し、糖鎖構造表記法、オントロジー、データベースなど糖鎖インフォマティクス研究に従事している。

氏名:新町 大輔
2010年に創価大学工学研究科にて修士号取得。2014年より創価大学の研究補佐員としてJSTの統合化推進プログラムに従事。現在は合同会社SparqLiteに異動し、セマンティックウェブ技術を用いた糖鎖インフォマティクスのシステム開発に従事している。

氏名:塩田 正明
2017年に創価大学工学研究科にて修士号取得。同年より創価大学の研究補佐員として統合化推進プログラムに従事し、エンジニアとして主に GlyCosmos Portal の開発・運用に携わっている。

氏名:三浦 信明
1998年北海道大学にて理論化学分野で博士号取得。いくつかの研究員を経て2003- 2012年は北海道大学特任准教授(2003-2008年度はサン・マイクロシステムズ寄付講座を担当)として糖鎖研究に従事。2013-2017年お茶の水女子大学勤務時にTotal Gylcome解析ソフト(TAG)の開発に着手(継続中)。現在は新潟大学特任准教授として人工知能を活用したメタプロテオームを中心としたバイオインフォマティクス研究に従事。

氏名:奥田 修二郎
2007年に京都大学大学院理学研究科にて博士号取得。京都大学化学研究所バイオインフォマティクスセンター博士研究員を経て、立命館大学生命科学部生命情報学科助教に就任し、生命科学系のデータベース開発およびマイクロビオームの研究を開始。2013年には新潟大学大学院医歯学総合研究科に准教授として異動し、医学分野のバイオインフォマティクスに従事している。

氏名:木下 聖子
1999年に米国ノースウェスタン大学よりコンピュータ工学の博士号を取得。台湾のアカデミアシニカ(中央研究所)のポスドクを経て、ロサンゼルスにあるBioDiscovery社にて上級ソフトウェア開発者として3年間勤務。2006年からは京都大学化学研究所バイオインフォマティクスセンターに移り、糖鎖インフォマティクス研究を開始。現在は創価大学教授として、教育と研究を続けながら糖鎖コミュニティのために便利な糖鎖インフォマティクスツールを開発し、生体システムにおける糖鎖の機能解明へ応用している。
本シリーズの第5回では、糖鎖のデータベースについて説明いたします。生体内には多様な構造を持つ糖鎖が存在し、遊離糖鎖をはじめ糖タンパク質や糖脂質などの複合糖質としても存在します。ここでは糖鎖のデータベースとして糖鎖構造および糖鎖抗原(糖鎖エピトープ)について、国際糖鎖構造リポジトリGlyTouCan、糖鎖構造のデータベースであるGlyCosmos Glycans、糖鎖局在のデータベースであるGlycomeAtlas、糖鎖の発現解析のデータベースであるTotalGlycome、糖鎖抗原・抗体のデータベースであるGlycoEpitopeを紹介します。
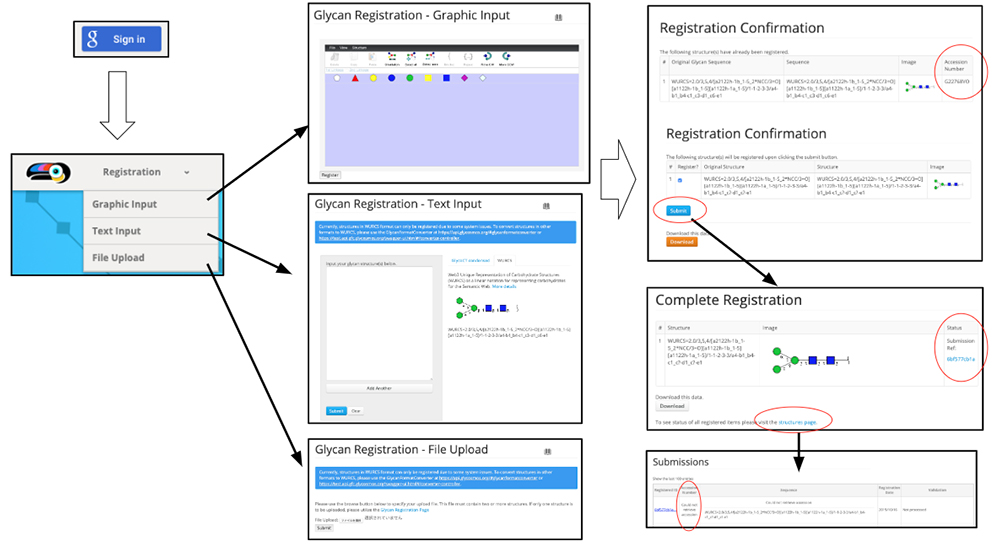
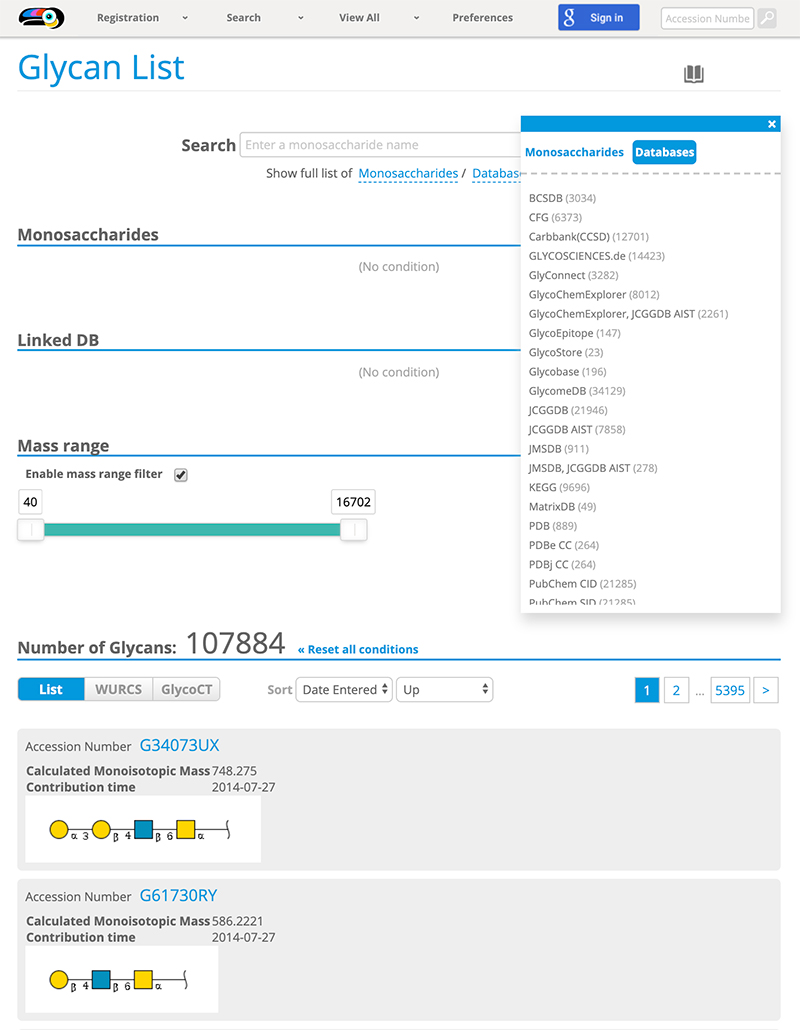
2013年に中国・大連において開催されたACGG−DB会議において、糖鎖科学研究において糖鎖構造を明確にする必要があることが議論されました。この議論により糖鎖構造のリポジトリを構築し糖鎖構造に固有のアクセッション番号を付与することが合意されました1。そして2014年8月に国際糖鎖構造リポジトリGlyTouCan 2が公開され、糖鎖構造に固有のアクセッション番号を付与することが可能となりました。GlyTouCanの初期のデータはGlycomeDB 3の糖鎖構造、生物種、他の糖鎖に関連したデータベースへのリンクなどの情報を引き継いで構築されました。現在(令和元年10月3日現在)では、115,802件の糖鎖の構造情報が収録されています。GlyTouCanはインターネット(https://glytoucan.org)を通して無料で利用することができます。糖鎖構造を登録したいユーザーは、はじめに“Sign in”する必要があります。その後、“Registration”メニューから糖鎖構造を3種類の方法(図 1)で登録することができます。糖鎖構造を入力し“submit”ボタンを押すと、「Registration Confirmation」ページへと遷移します。ここですでに登録されている糖鎖構造の場合は、右端にアクセッション番号が表示されます。表示されない場合は登録されていない構造になります。入力した構造を確認し、“Submit”ボタンを押しますと、「Complete Registration」ページへ遷移します。ページ下方の青い“structures page”をクリックすると、ユーザーから受付けた糖鎖構造情報の一覧が表示されます。これらの受付けた糖鎖構造は、GlyTouCanのシステムにより1時間毎に登録処理が行われます。このページのテーブルにある2列目の“Accession Number”欄の“Could not retrieve accession”に“G”ではじまるアクセッション番号が表示されますと、受付けた糖鎖構造にアクセッション番号が付与されたことになります。これにより、登録した糖鎖構造のアクセッション番号が利用可能となります。また、すでに登録されている糖鎖構造を検索したい場合は、“Search”メニューや”View All”メニューを利用することができます(図 2)。詳しい利用方法などについては、“User Guide”に記載してあります。
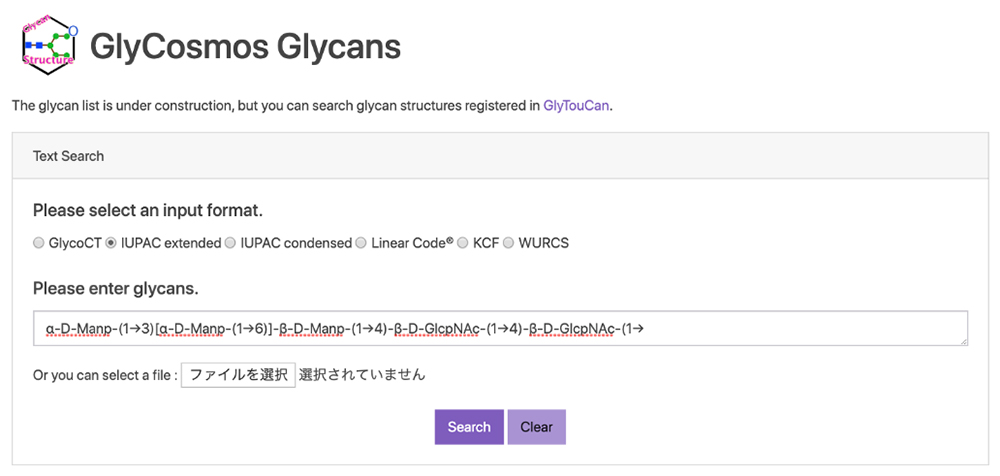
GlyCosmos Glycansは、GlyTouCanが糖鎖構造の登録に特化したことにより、生物種、文献、外部データベースへのリンクなどを公開するために開発されています。現在(令和元年10月16日現在)は、文字列形式の検索が利用可能で、6種類の糖鎖構造形式(GlycoCT 4, IUPAC extended 5, IUPAC condensed 5, Linear Code® 6, KCF 7, WURCS 8)が利用できます(図 3)。主な形式は本連載の糖鎖命名法及び糖鎖関連リソースの概要において説明されています。
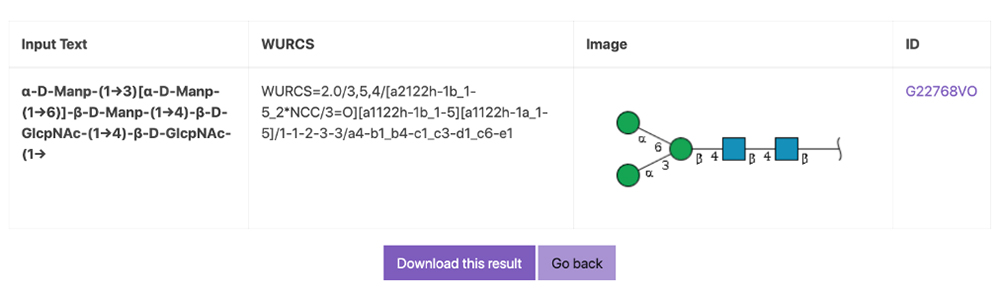
検索結果が表示され右端のID欄が“Not Found”と表示された場合、検索クエリーの糖鎖構造がデータベースに登録されていないことがわかります(図 4)。

また、“G”ではじまるアクセッション番号が表示された場合、検索クエリーの糖鎖構造がデータベースに登録されています(図 5)。ここで、リンクをクリックすると目的の糖鎖構造のページが開きます。現在はGlyTouCanのエントリーページにリンクされています。今後、生物種、組織や生物種別糖鎖構造の一覧、単糖リスト、他のデータベースへのリンクなどの情報にアクセス可能なサイトを開発する予定です。
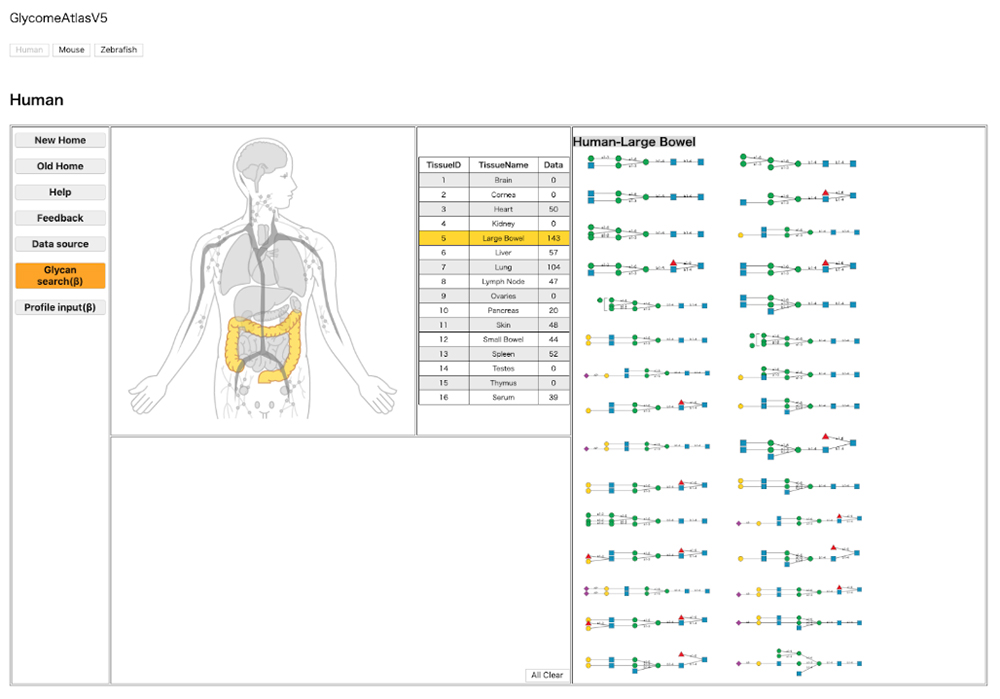
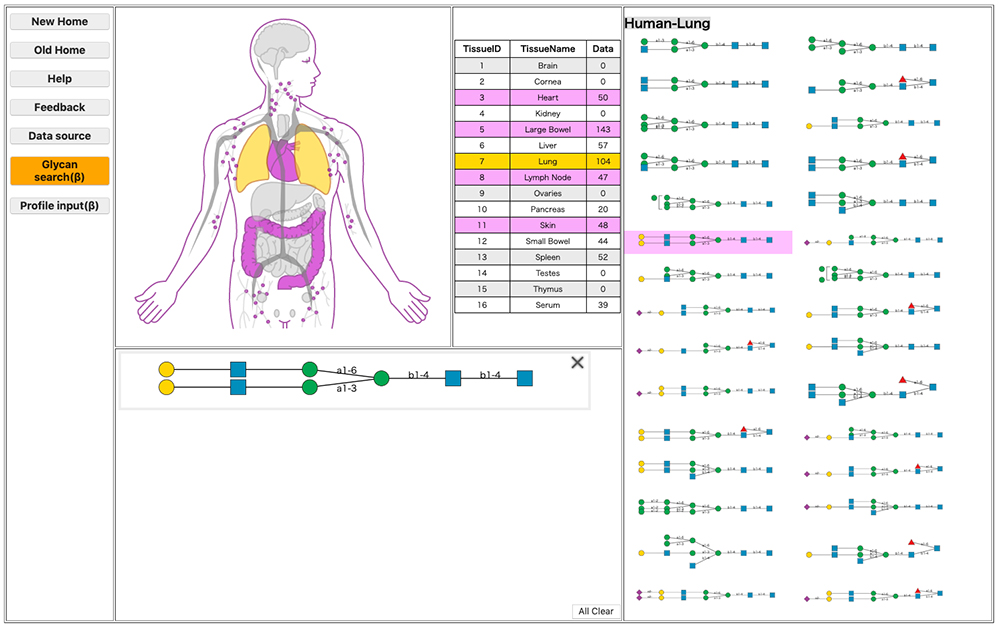
糖鎖はその局在部位と連動して生体内での構造が次々と変化しています。これらの構造変化や局在部位の変化は、糖鎖の機能発現と密接に関係しています。そのため、糖鎖の局在を把握することは糖鎖科学研究において重要となります。GlycomeAtlas 9は生体内における糖鎖の局在を閲覧することができます。現在(令和元年10月30日現在)は、ヒト、マウス、ゼブラフィッシュにおける糖鎖構造の局在を閲覧することができます。ヒトとマウスについてはCFG(Consortium for Functional Glycomics)Glycan Profilling data、ゼブラフィッシュは文献 10のデータを利用しています。
ページ上部の「Human」、「Mouse」、「Zebrafish」のボタンをクリックすると生物種を変更することができます。そこで、Tissueをクリックすると該当する組織がハイライトされ、含まれる糖鎖構造が右側に表示されます(図 6)。また、右の欄に表示されている糖鎖構造を選択すると、その糖鎖構造の局在部位がハイライトされます(図 7)。
糖鎖は、生体内で病原体の侵入やこれに対抗する免疫に関連した分子認識による細胞間コミュニケーション、シグナル伝達を通した生命機能の制御、タンパク質生合成のホールディング支援と品質管理および局在の指示となるタグ付けといったような膨大な情報を制御しています。
糖鎖の発現量は、疾患などの生命現象によって変化すると言われていて、その変化を追跡する研究は古くから盛んに行われておりました。従来の研究では測定法をはじめとした様々な理由から、特定のクラスの1個から一群の糖鎖(サブグライコーム)にフォーカスして変動を観察する方法が主流でした。近年の質量分析技術の進歩や新しい分析手法の発展によって異なるクラスの糖鎖群を包括的に分析できるようになり、総合グライコーム(Total Glycome)として注目されています11, 12。Total Glycomeでは現在N-結合型糖鎖(N-glycan)、O-結合型糖鎖(O-glycan)、糖脂質糖鎖(GSL-glycan)、グリコサミノグリカン(GAG)、遊離糖鎖(fOS)という5つのクラスの糖鎖を網羅的に定性・定量分析することができます。いっぺんに200を超える糖鎖の発現パターンを疾患や分化などの生命現象におけるグライコーム追跡可能な方法です。Total Glycomeを調べる意味としては
● 細胞表面には様々な複合糖質があり全体の発現をマーカー探索などに利用できること
● 共通のエピトープが様々なサブグライコームに存在すること
● ある糖鎖の合成不全を別なクラスの糖鎖が補償している可能性があること
● N型糖鎖と遊離N型糖鎖の代謝経路は密接に関連していること
などがあげられます。Total GlycomeのデータベースはGlyCosmosポータルの1データベースとして位置付けられ、今後膨大なデータを得るであろうTotal Glycomeを俯瞰的に観察し他オミックスとの連携の目的で構築されました。
Total Glycome Databaseは、N-glycan、O-glycan、GSL-glycan、fOS、GAGの発現解析結果をデータベース化し様々なビューを提供しています。データベース画面上部の “model”, “source”, “select a view”, “glycan class”のプルダウンメニューから、各項目について選択し、“Submit”ボタンを押すことにより、選択したデータセットが可視化されます。現在は、表形式でのビューをはじめ棒グラフによるそれぞれの糖鎖発現量の比較や円グラフによる各クラスの糖鎖発現割合の横断的な変化等のビューを用意しています(図 8)。
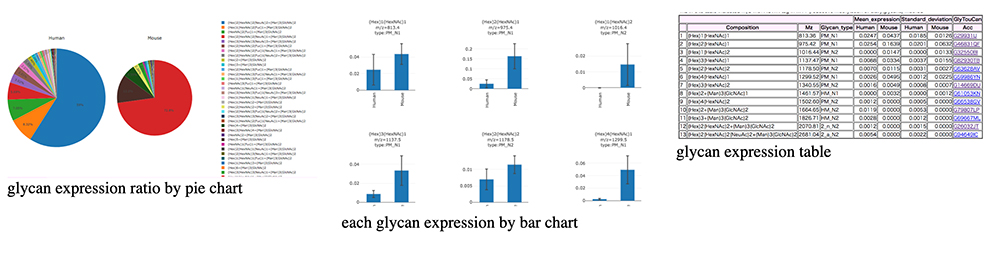
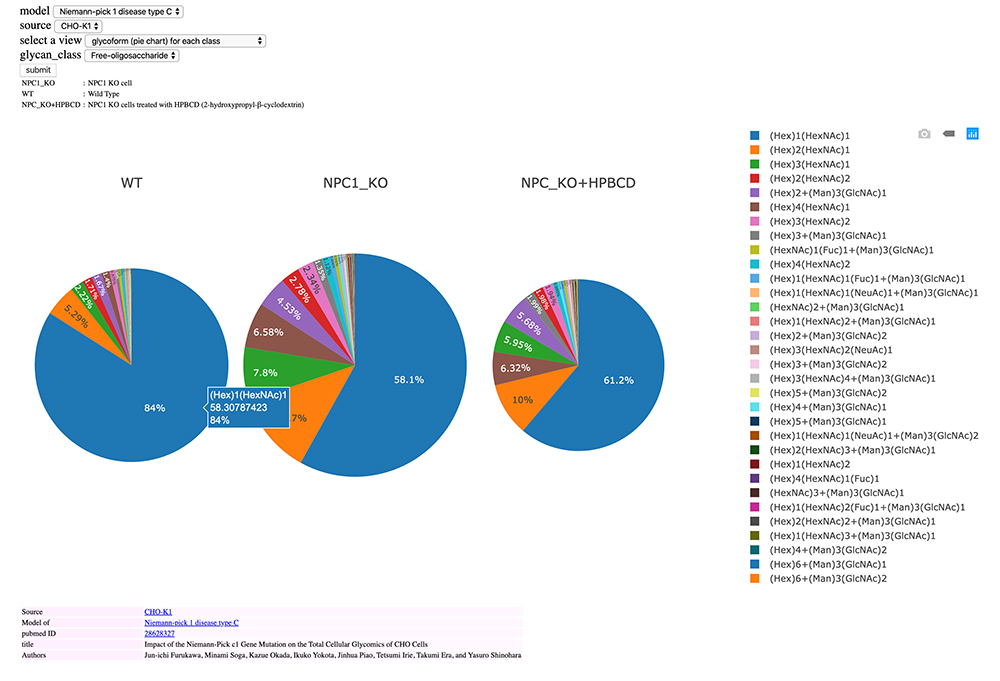
図 9はNiemann-Pick病C型(NPC)のモデル細胞(野生型CHO細胞、NPC1遺伝子をノックアウトしたものさらに、NPCに対する有効性が報告されている2-hydroxypropyl-β-cyclodextrin(HPBCD)を添加したものの3種)のGlycome解析結果13のうちfOSの発現量を円グラフで表示したものです。円の大きさがfOS全体の総発現量を示します。ここで円グラフにマウスオーバするとカラーごとに糖鎖構造等の情報が表示されます。図 9では最も発現割合が大きい (Hex)1(HexNAc)1 が表示されているのが見て取れます。NPC1遺伝子のノックアウトによって糖鎖発現量が増加しますが (Hex)1(HexNAc)1 の割合は著しく減少します。治療薬モデル化合物HPBCDの投与によってfOSの発現量は減少し野生型CHO細胞の発現量に近づきますが (Hex)1(HexNAc)1 の割合は増加するものの、それほど回復しないことが見て取れます。このような発現変動の検出法は治療薬候補を多く用意して同様の実験を多数行う事によってグライコームに基づいた治療薬のスクリーニングへと展開できる可能性も考えられます。それには本データベース で示したような容易に可視化を行えるシステムが有用であるといえます。
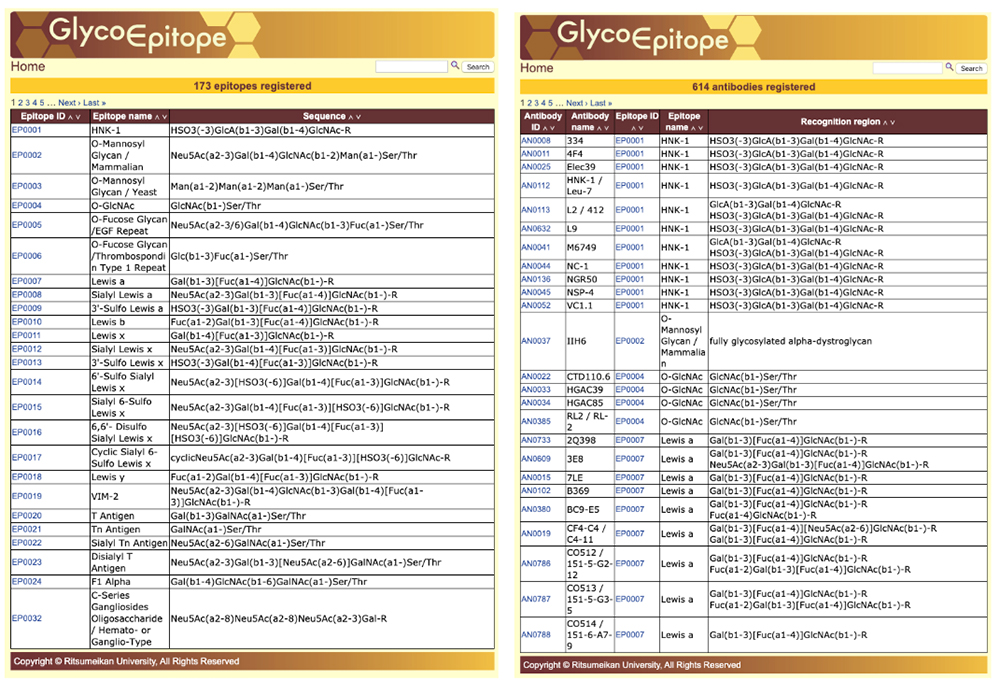
GlycoEpitope 14,15,16は糖鎖抗原と糖鎖抗原を認識する抗体についての情報を収集したデータベースです。糖鎖生物学者の協力を得て構築されています。このデータベースはGlyCosmosとhttps://glycoepitope.jpから利用することができます。GlycoEipopeはエピトープの一般データ(General)、エピトープを認識する抗体(Antibody)、発現しているタンパク質(Glycoprotein)、エピトープ部分構造を持つ糖脂質、エピトープの合成や分解に関与する酵素、食物糖鎖、文献から構成されています。現在(令和元年10月30日現在)で、173のエピトープ、614の抗体の情報が収録されています。
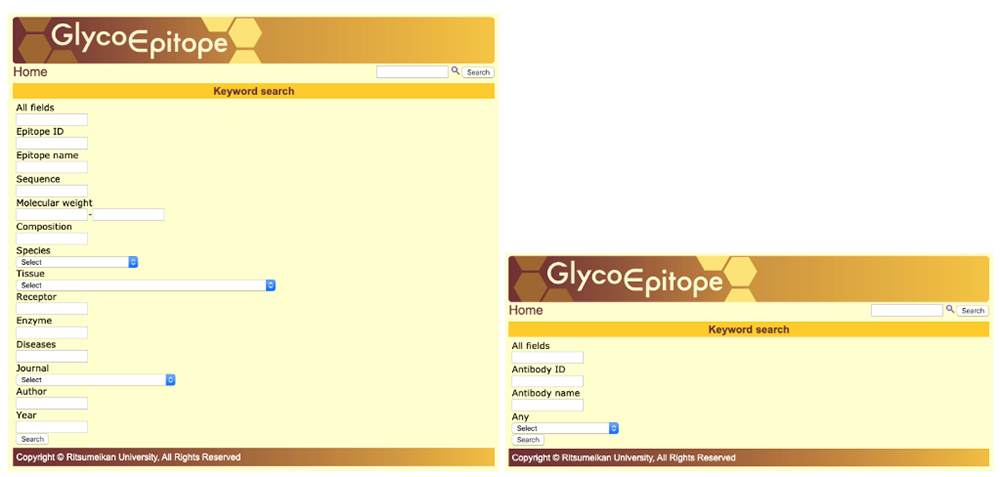
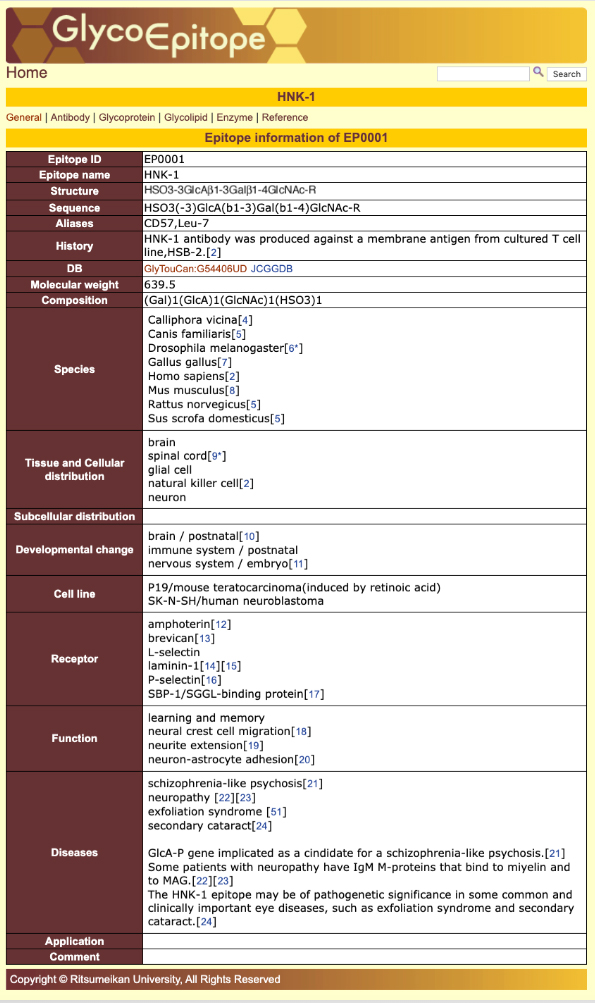
糖鎖抗原や抗体の一覧表示(図10)や各項目のキーワード検索(図11)から目的の情報を検索することができます。糖鎖抗原の詳細ページでは、各種情報を閲覧することができます(図12)。