
氏名:木下 聖子
1999年に米国ノースウェスタン大学よりコンピュータ工学の博士号を取得。台湾のアカデミアシニカ(中央研究所)のポスドクを経て、ロサンゼルスにあるBioDiscovery社にて上級ソフトウェア開発者として3年間勤務。2006年からは京都大学化学研究所バイオインフォマティクスセンターに移り、糖鎖インフォマティクス研究を開始。現在は創価大学教授として、教育と研究を続けながら糖鎖コミュニティのために便利な糖鎖インフォマティクスツールを開発し、生体システムにおける糖鎖の機能解明へ応用している。
本シリーズの最終回では、糖鎖関連データベース開発にかかわる様々な取り組みへの人工知能(AI)の活用の可能性について述べます。 AI領域の簡単な紹介の後に、糖鎖生物学とAIの適合性について記載し、続いてこれらの領域を統合した将来の研究について考えを述べます。
「人工知能(AI)」という用語は、ロボット工学1やWebシステム2、ユビキタスコンピューティング2,3から、機械学習・ディープラーニング4に至るまで、幅広い研究トピックを内包しています。AI領域の中でも特に糖鎖生物学と関連が深いのは、機械学習、そしてディープラーニングです。
現在、機械学習では、学習の種類(教師あり、教師なしなど)やデータセット(分類、パターン予測など)に応じて様々な方法を利用できるようになっていますが、一般に機械学習とは、大量のデータを特定のモデル(テキスト、グラフ、画像など)の下で処理し、データ内において確率の高いパターンを「学習」しようとするプロセスを踏みます。この学習プロセスは、いくつかの共通点を含むことがわかっているデータセットを用いて「学習」を試みるとともに、モデルのパラメータを繰り返し調節し、多くの場合「コントロール」となるデータセットと比較することで、ターゲットデータについて高い確率(または「精度」)で予測を行うことができるようにするものです。真陽性と偽陽性を区別することが難しいモデルも多く、このプロセスは必ずしも簡単には行きません。また、モデルを過剰に学習してしまうことで、学習データに特異的すぎて他のデータセットに一般化できないパターンをモデルが学習してしまう危険性もあります(オーバーフィットと呼ばれます)。しかし、あるモデルについて高い精度で一般化できることが示されれば、そのモデルは「学習済み」であると考えることができ、これを用いて「未知のデータセット内に学習されたパターンが含まれているか」や、「未知のデータセットを特定のカテゴリに分類できるか」といったことを「予測」することができます。この予測は通常、確率(あるいは尤度)の出力という形で行われます。
このような機械学習モデル(およびほとんどの人工知能モデル)におけるボトルネックの1つは、訓練されたモデルがブラックボックスであることが多く、学習結果の実質的な「意味」がわからないという点です。近年では、このブラックボックスの中身を明らかにしようという取り組みがなされています(学習のホワイトボックス化、解釈可能な機械学習など)5。この本に書かれているように、決定木のような解釈可能なモデルを使用することはもちろん可能ですが、これらは必ずしも強力な方法であるとは限らず、未知のデータを含む大規模なデータセットに対して一般化できない場合もあります。一方、学習したモデルのブラックボックスを透明化し、学習したデータの特徴を明らかにする1つの方法として、「特徴抽出」と呼ばれる処理があります。データセットから学習した実際のパターンを実験者にフィードバックできるようにするためのこのような技術は、生命科学において重要になるでしょう。実験者が予測結果をより簡単に検証できるようになるからです。しかし、このような技術は、機械学習の分野では未だ研究の余地がある領域であり、糖鎖情報学においてはまだ黎明期にある分野です。
ところで、糖鎖生物学には、曖昧さの中に意味を見出そうとするプロセスがあるともいえます。この曖昧さというのは、糖鎖の構造に曖昧さがあるということです。糖鎖の構造は、他の重要な生体分子(タンパク質など)に比べてかなり柔軟であることが知られているほか、構造単位の繰り返しや多種多様な修飾(例えば、硫酸化、リン酸化など)が存在します。また、レクチンや酵素による認識にも曖昧さがありますが、これもその多くが糖鎖の構造的な曖昧さに起因するものです。
糖鎖生物学にはこのような曖昧さがあるため、人工知能や機械学習の手法を応用する対象として適した分野であると考えられます。一般的に、実験データをスクリーニングして、実験プロトコルにおける次段階の選択肢を絞り込む際には、バイオインフォマティクスのツールがよく使われています。BLASTはこのようなツールの一例であり、数十億という配列を数分間でスクリーニングすることが可能です。このように、バイオインフォマティクスデータベースは、ハイスループットな実験技術によって生成された大量の実験データの保存・整理に有用です。機械学習や人工知能技術は高品質なデータを大量に必要とするため、結果としてこのようなデータベースが、解析に欠かせないものとなります。糖鎖生物学のデータとしては、糖鎖の曖昧さや認識性、結合親和性を記述したデータを格納し、そこから意味を抽出できるようにすることが重要です。これは、確率や統計学を利用し、さらにそのような情報を内包できる標準化された用語を用いることで可能になります。また、「Garbage in, garbage out(低質なデータを入力すると、低質な結果しか得られない)」という言葉があるように、データについて効果的な学習を行って価値ある情報を抽出するには、高品質で精選されたデータを格納しておくことも大切です。そのため、データそのものとともにデータの質を確実に記録するためにも、標準化が重要になります(MIRAGEに関して解説した本シリーズの第2回をご覧ください)。実際にGlySpace Allianceでは、アライアンス全体で品質管理が確実に行われるような合意がなされています6。
セマンティックウェブ(SemWeb)技術は、標準化した意味をデータとともに格納する強力な方法の1つであり、その生命科学への応用については過去の論文で解説しています7。端的に言うと、SemWebではオントロジーを使用しますが、、これは管理された語彙とそれらの関係性を形式的に定義したものです。よく知られている例としてはジーン・オントロジー(Gene Ontology)8があります。これは、すべての用語に識別子を与え、遺伝子の機能、細胞内での局在、分子の特性などを階層的に定義したものです。そしてSemWebでは、このようなオントロジーに関するターム(term)とともにデータを格納することで、データに意味を付与できるようになっています。図1に、オントロジーとデータベース、そしてこれらの関係性を図示しました。オントロジーは、データに内包される概念を記述するためのタームで構成されています。そして、これらのタームは互いに関係性を持っています。例えばこの図では、Term AとTerm Bは、小さな違いはあるかもしれませんが、同等の概念を表しています。一方でTerm Cは全く別の概念であり、Term A(およびTerm B。Term Aと同等であるため)と何らかの関係を持っています。このようにオントロジーには、タームやそれらの関係性が多数定義されています(図中のTerm X)。次に、この図の右側に示したように、データベースには、はじめはコンピュータで解釈できないデータ(灰色で示したデータ)が含まれています。つまり、データ間に何の関係性もない状態で別々に格納されています。しかし、このデータには、これらを記述するためのオントロジータームとして、メタデータを付与することができます。図に示したように、Data1とData2はTerm A、Data3はTerm B、そしてData4はTerm Cの概念として定義されています。この追加情報が付与されていることで、オントロジーで定義した関係性を、結果的にデータの中で推測することができるようになります。つまり、Data1とData2(全く同じ概念)の関係性や、Data3とのつながり(Data1およびData2と同等)、さらにはData4が他の3つのデータすべてと何らかの形で関係していることを、人間とコンピュータの両方が推測できるのです(黒矢印および等号)。
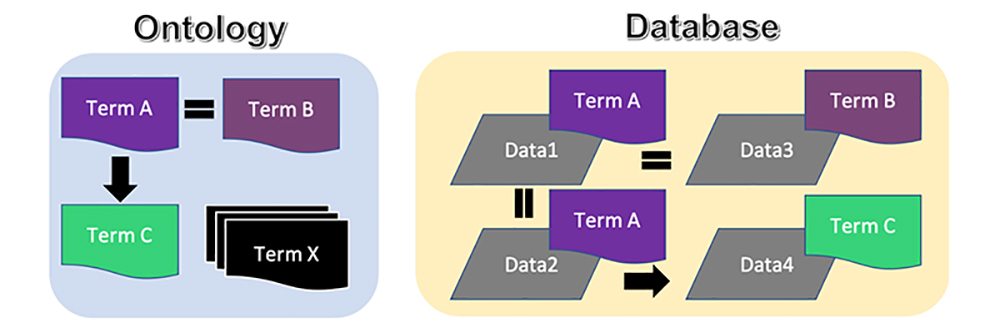
データベースをまたいで使用できるような標準化されたオントロジーを作成することで、データに付与されるこれらの意味を結果として標準化できるため、データセットを容易に統合することが可能になります。つまり、SemWebの使用には、(1)SPARQLのクエリ言語を使用することで、異なるサーバーやネットワークにまたがったバラバラのデータセットに対して、統合されたデータセットとして検索ができ(2)インターネット上の様々な場所にあるデータから新しい関係性を推測するようコンピュータを学習させることができる、という利点があります。つまり、SemWeb技術を実装することで、これまで「島」と呼ばれていたデータ9を統合でき、これらのデータに対して一括で検索ができるようになるのです。
SemWeb研究における最近の進歩としては、さらに類似性関数の発展も挙げられます10,11。0と1をベースとしているコンピュータにとって、「類似性」という概念を扱うことは通常困難です。しかし、セマンティック分析やオントロジーを利用することで、データ間の相関性や類似性を推定できることが示されています10。つまり、このような関数の糖鎖データへの応用が期待でき、セマンティックウェブ上の関連データから曖昧さの概念を保存、処理、さらには学習することができるようになるかもしれないのです。
オントロジーを開発し、このオントロジーを用いてデータベースを構築し、さらにこの糖鎖生物学的データから学習を行ってコンピュータモデルや手法を実装できるようになるまでには、まだまだ長い道のりが待っていますが、今後の糖質科学研究に開かれている道筋を垣間見ることはできます。例えば、タンパク質ファミリーをまたいで糖鎖修飾のパターンを抽出することができることが示されています。UniProt 12といった主要なタンパク質データベースへのタンパク質糖鎖修飾情報の蓄積に伴って、タンパク質の糖鎖修飾部位を予測できる機械学習手法も開発されてきました13-15。このような手法を、Protein Data Bank(PDB)の3次元データを用いてさらに拡張することで、例えば基質の構造パターンをより深く理解したり、真核生物以外の種に応用したり、といったことも可能になります。もし糖鎖アレイ実験などで得られた糖鎖結合パターンデータのフォーマットを、実験技術を超えて標準化・整理できるようになれば、近い将来、生物種間でこれらの相関関係を解析できるようになるかもしれません。過去10年間の糖鎖アレイ技術の進歩によって16-21、糖鎖を認識する特定の糖鎖結合タンパク質、ウイルス、細菌などについてのデータが大量に生成されています。また、このようなデータを解析するためのソフトウェアツールが多数利用できるようになりました。しかし、糖鎖結合パターンについて標準化およびキュレーションがなされたデータベースは存在しません。AIを応用して糖鎖機能を理解する上で、このステップは重要です。さらに、このような糖鎖アレイデータを、結合パートナーの3次元構造と統合することで、糖鎖の2次元および3次元構造の認識プロセスの解析に機械学習を利用できる可能性もあります。
長期的には、このようなオミクスデータをすべてGlyCosmosに統合することで、システム糖鎖生物学の研究に利用することができます。このデータベースを活用できる研究分野は、(1)糖鎖生合成のシミュレーション、(2)糖鎖結合による細胞シグナリングのシミュレーション、および(3)細胞外基質(ECM)のシミュレーション、の3つに大きく分けることができます。(1)については、AIを用いて糖転移酵素の基質特異性を予測することが可能です。糖鎖生合成シミュレーションにおいてボトルネックとなっているのは、基質特異性の定義と、酵素反応が生じるために必要となる基質への制約です。特異性や酵素の特性がすでに知られている糖鎖遺伝子のデータベースを用いることで、ここで挙げたような予測を行うためのモデルを学習することができます。また同様に、糖転移酵素の反応パラメータも得ることが難しいですが、既知のデータに対して機械学習を行うことで、詳細なシミュレーション解析に必要なKmやVmaxといった反応パラメータの値を予測できます。
細胞シグナリングとECMのシミュレーション研究については、目的達成のために利用できるシステム糖鎖生物学モデルが、今のところほとんど存在していません。糖鎖生物学の領域ではよく知られていることですが、シグナル伝達は糖鎖修飾による調節を受けていることが多く22-26、リン酸化と競合してシグナル伝達を調節するタイプの糖鎖修飾も実際に報告されています27。このような糖鎖に関連したシグナル伝達経路のデータベースは基本的に存在しておらず、シグナル伝達のパラメーターについても無論まとまった情報がありません。したがって今後は、このようなデータを蓄積するとともに、関連するオミクスデータセットとの統合作業が必要となり、これによってシミュレーションの実行とシグナル伝達パラメータの予測が可能になるでしょう。ECMにおける糖鎖とその機能については、理解が深まってきてはいるものの28、ECM内での糖鎖構造の詳細(特にプロテオグリカン)や、他のタンパク質との相互作用、およびこれらの立体的な相互作用の詳細など、形式的に表現しなければならない点がまだたくさんあります。この草分けとなったのが、ネットワーク解析用のツールも提供しているMatrixDB 29であり、今後ECMに関するシステム糖鎖生物学研究をさらに進めていく上で、重要なデータベースになるでしょう。
最後に、このシリーズの前稿「糖鎖情報科学の協働体制」でも紹介しましたが、システム糖鎖生物学研究のためのデータベースやツールの開発を目的として、システム糖鎖生物学コンソーシアム(SysGlyco)が結成されました。現在SysGlycoは、GlycoSimやGlycoEnzDBといったツールを開発している研究者で構成されています。これらのツールもまた、システム糖鎖生物学のデータを標準化して整理するために必要な関連情報を、さらに深化・統合するための足がかりとなるものです。
まとめると、糖鎖科学のデータにAIやSemWeb技術を応用することで糖鎖の機能をよりよく理解するという具体的な目標に向けて、多くのエキサイティングな研究が進められています。このような研究では必ず、多様なバックグラウンドや専門性を持つ研究者同士の協力が必要です。AIを利用するにしても、実験科学者しか得ることのできない大量のデータが必要となります。異なる研究分野間には専門用語の壁が存在するため、協力体制を築くことは容易ではありません。しかし、このような共同研究が成功すれば、その成果は広範囲に影響力が及ぶものとなる可能性があります。現在は医療分野に注目が集まっていますが、農業やエネルギーといった分野も糖鎖科学との間に結びつきがあります30。共同研究を進めていくことで、糖鎖科学の世界がこのような分野にまで拡がっていくことができるのではないかと、個人的には期待しています。