
氏名:山田 一作
1997年に東京都立大大学院工学研究科にて博士号取得。名古屋大学(日本学術振興会特別研究員(COE))などを経て、2002年から野口研究所研究員、2006年から同所にて糖鎖科学研究を開始し、糖鎖構造表記法、オントロジー、データベースなど糖鎖インフォマティクス研究に従事している。

氏名:木下 聖子
1999年に米国ノースウェスタン大学よりコンピュータ工学の博士号を取得。台湾のアカデミアシニカ(中央研究所)のポスドクを経て、ロサンゼルスにあるBioDiscovery社にて上級ソフトウェア開発者として3年間勤務。2006年からは京都大学化学研究所バイオインフォマティクスセンターに移り、糖鎖インフォマティクス研究を開始。現在は創価大学教授として、教育と研究を続けながら糖鎖コミュニティのために便利な糖鎖インフォマティクスツールを開発し、生体システムにおける糖鎖の機能解明へ応用している。
本シリーズの第8回では、個々の研究者、研究室、プロジェクトでは解決が難しい糖鎖科学の課題に対して協働体制を組織化して課題に挑戦するための協力的および組織的な取り組みについて紹介します。
これまで様々な国で糖鎖関連の研究開発プロジェクトが実施され、その中の糖鎖インフォマティクス分野において各種データベース等が開発されてきました。しかし、これらのプロジェクト終了に伴って、開発されたデータベース等がウェブサイトの閉鎖などにより利用することが困難になり、糖鎖研究において障害となりつつありました。この問題を解決するために、世界の糖鎖インフォマティクス研究者が協調・連携しデータベースや各種ツールなどを共同で開発する仕組みを検討し、糖鎖インフォマティクスコンソーシアム(Glycoinformatics Consortium)を野口研究所の支援のもと設立するに至りました。
糖質情報学のための情報を一元化したソフトウェアリポジトリの提供および管理、そして糖質情報学・糖質化学・糖質生物学のための情報交換の場を設け、糖質科学の発展に貢献することです。
糖鎖関連データベースや各種ツールのカタログ作成やソフトウェアリポジトリの提供・管理により、研究者が利用しやすい環境を整備しています。また、use case募集ワークショップやハンズオンセミナーの開催をしています。これらの活動はウェブサイト(https://glic.glycoinfo.org)で公開しています。
糖鎖表記法、オントロジー、リソース・ディスクリプション・フレームワーク(Resource Description Framework )のワーキンググループを組織し、GLICソフトウェアリポジトリの推進やソフトウェアやデータベースの信頼性についての検討、糖鎖表記法のドキュメントの整備や検証法の検討などを実施しています。そして、ウェブサイトにおいてドキュメントの公開や注意点などを公開しています。また、糖鎖科学研究に関するuse caseワークショップを開催し、集計・整理し糖質科学の発展に貢献しています。
CarbBank 1の開発以来、糖鎖関連のデータベースが大量に開発されており、それぞれに糖鎖構造、局在、および機能に関する独自のメタデータセットが格納されています。糖鎖構造リポジトリGlyTouCan 2の開発は、糖鎖構造のデータを整理して研究者やデータベース間で糖鎖情報をより簡単に共有できるようにするのに大いに役立ちました。そして、2018年に3つの主要な糖鎖関連データベース統合プロジェクト(スイスのGlycomics@ExPASy 3、米国のGlyGen 4、および日本のGlyCosmos 5)が開始されました 。これらのプロジェクトが研究開発を重複させないようにするために、これら3つのグループで構成されるGlySpace Allianceが2018年8月に設立されました6。
GlySpace Allianceの包括的な目的は、生物学的に関連のある糖鎖の構造、それらが含まれる複合糖質、それらの生合成、および生物学的プロセスにおけるそれらの役割に関する信頼できる情報を科学コミュニティに提供することです。
アライアンスのメンバーは糖鎖関連のマルチオミクスデータ統合プロジェクトであり、データベースの開発、保守、および普及のための資金を確保している必要があります。さらに、各メンバーは、アライアンスに価値のあるデータを提供し、ユーザーのニーズを評価するためのデータ使用に関するフィードバック(アクセスログなど)を提供し、アライアンス会議に出席する必要があります。
会議は対面式で年に1度開催しますが、必要に応じてオンライン会議を開催します。会議では、データの共有や標準情報について協議します。主要なデータ標準に従う必要があり、例えばタンパク質配列にはUniProtKB 7またはRefSeq 8を使用し、糖鎖構造にはGlyTouCan 2、PubChem 9またはChEBI 10を使用します。
GlySpace AllianceのTraining sessionも計画しており、メンバーのそれぞれのポータルの利用方法を教授し、ハンズオン形式でユーザーの個別のデータを用いて解析する方法を演習します。詳細はGlySpace Allianceのホームページに掲載します。
塩基配列共同体のINSDCやアミノ酸配列データベースコンソーシアムのUniProt、またタンパク質の立体構造データベースwwPDB同様、世界中の糖鎖関連データベースを共有する仕組みがGlySpace Allianceとして設立されました。本アライアンスが設立されたことで、世界中の糖鎖関連データベースが統一化されると期待できます。

システム糖鎖生物学コンソーシアム(https://sysglyco.org/)の目的は、グリコシル化(グリカン生合成)だけでなく、シグナル伝達経路、遺伝的相互作用など必要とするシステム糖鎖生物学研究のためのオープンプラットフォームを開発することです。
具体的には、コンソーシアムは以下を収集して提供します。
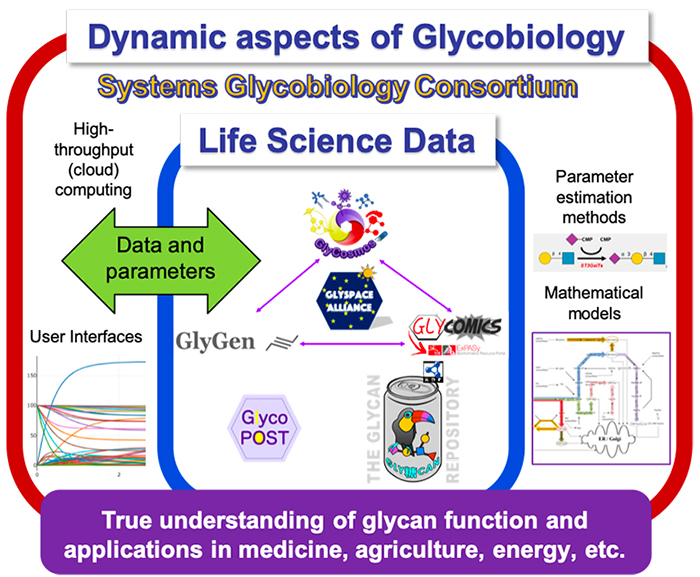
2019年末に設立された本コンソーシアムはまだ準備段階にありますが、メーリングリストを通して世界中のシステム糖鎖生物学領域の研究者が交流できる場を提供します。数理モデルに必要な多くの反応パラメータやモデルをデータベース化し、基質となる糖鎖の標準形式などが決定されれば、糖鎖の機能解明を網羅的に解析できるようになると期待できます。