
氏名:鈴木龍一郎
秋田県立大学生物資源科学部生物生産科学科助教
山形大学で長谷川典巳教授および久野敦助手の指導の下、キシラナーゼの機能改変の研究で2005 年に博士(理学)を取得。2005 年から平林淳博士(産業技術総合研究所)の下でR型レクチンの構造機能相関の研究に従事した後、2006 年からは伏信進矢教授(東京大学)の下でミルクオリゴ糖代謝関連酵素の構造と機能の研究に従事。その後、舟根和美博士(食品総合研究所)の下で環状イソマルトオリゴ糖代謝関連酵素の機能改変の研究に従事(2010年–2012年)。2012年より現職。研究の関心は、α-グルカン代謝関連酵素の構造と機能の解明と、その成果のα-グルカンの酵素合成への応用にある。

氏名:鈴木英治
秋田県立大学生物資源科学部生物生産科学科教授
埼玉大学理学部生化学科卒業(1985 年)。宮地重遠教授の指導の下、1990 年に東京大学大学院で理学博士を取得。茨城大学理学部生物学科助手(1990 年–1997 年)、地球生命環境科学科助教授(1997 年–2001 年)を務めた。2001 年に秋田県立大学生物資源科学部生物生産科学科に助教授として着任、2006 年から准教授、2011 年から現職。研究の関心は、光合成生物、特にシアノバクテリアにおける炭水化物代謝の生理学、多様性および進化にある。
澱粉は植物が光合成産物として生産する多糖であり、穀類、芋類、および豆類など人類の主食である身近な食品に含まれている。澱粉は食品・食品添加物にとどまらず、糊やバイオプラスチックなどの素材としても工業的に利用されている。澱粉は規則正しい分岐構造から成るアミロペクチンを主成分としており、アミロペクチンの構造は食味・物性に影響を与えることがわかっている。アミロペクチンはスターチシンターゼ、枝作り酵素、枝切り酵素など複数の酵素群によって生合成されるが、構造を制御する仕組みはわかっていない。アミロペクチンの構造を制御する仕組みを明らかにすれば、意図した構造・性質のアミロペクチンを作れる可能性がある。これまでに著者らは、枝作り酵素が生産する分岐鎖の長さを制御する仕組みを明らかにしてきた。本稿ではアミロペクチンの構造と性質、構造を制御するために解決すべき課題、および枝作り酵素の研究の現状について概説する。構造を制御したアミロペクチンの生産が可能となれば、食料自給率の向上および脱炭素化などに繋がり、わが国の発展に貢献すると期待される。
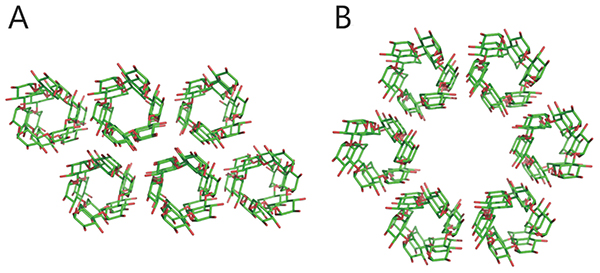
貯蔵多糖(分岐 α-グルカン)には澱粉およびグリコーゲンが存在し、それぞれの構造と性質は異なっている。澱粉はアミロースおよびアミロペクチンから成り、アミロペクチンを主成分としている。アミロースは、数百から数万のグルコースが α-1,4-結合で連結した多糖である1。α-1,4-グルカン直鎖は分子内水素結合を形成し、らせん状となる。アミロースは α-1,6-結合から成る分岐点をわずかに含んでいる(割合は1% 未満)が1,2、ほぼ直鎖状であるため、らせん状の立体構造をとる(図 1A)。アミロペクチンは数万のグルコースが α-1,4-結合で連結したグルカン直鎖に、α-1,6-結合から成る分岐点を介した分岐鎖が5–6% の割合で付加した構造をとる1。アミロペクチンは、複数の房(クラスター)がタンデムに連結したタンデムクラスター構造をとると考えられている1,3,4(図 1B)。この構造は、分岐点が多い領域と少ない領域の繰り返しから成っており(図 1B)、分岐点が少ない領域は分岐鎖同士で二重らせん構造を形成できる(図 1C)。らせん構造を形成すると水分子が排除されて結晶性が生じ、不溶性となる。このことから分岐点が多い領域および少ない領域を、それぞれアモルファスラメラおよびクリスタルラメラと呼ぶ1,3,4。クリスタルラメラのパッキングはA形およびB形が知られており、A形結晶およびB形結晶には、それぞれ穀類および芋類の澱粉などが含まれる1(図 2)。さらに、A形とB形の混合型も存在し、C形と呼ばれる1。
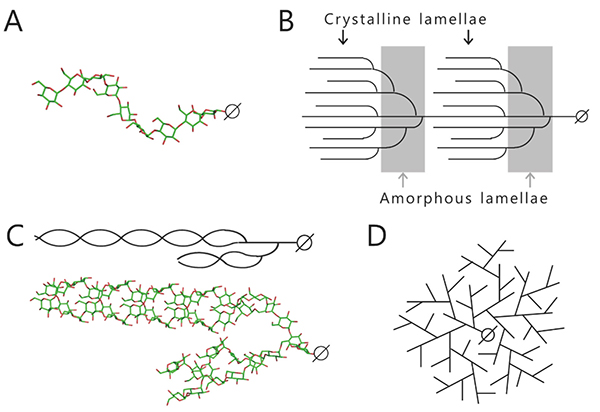
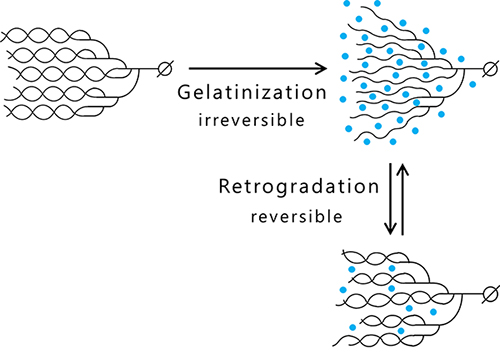
アミロースおよびアミロペクチンはヨウ素で染色されるが、これはヨウ素分子がらせん構造の内部に入り込むことで起こる。長鎖から成るアミロースは青紫色に染色されるが、短い分岐鎖を含むアミロペクチンは赤紫色に染色される。 アミロースおよびアミロペクチンは、加水・加熱処理によってらせん構造がほどけて水分子が入り込むことで糊化(α化)し、粘性を示すようになる(図 3)。これを冷却すると大部分の水分子が排除され、らせん構造を再形成することで老化(β化)し、硬くなる4(図 3)。長い直鎖状のアミロースは離水しやすいため、老化しやすい。一方、アミロペクチンはアミロースよりも短い分岐鎖を持つため、老化しにくい性質を示す。
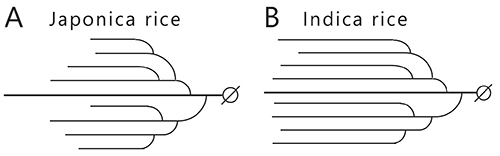
アミロペクチンおよびアミロースの性質が米の食味に与える影響について紹介したい。イネの栽培種にはジャポニカ種やインディカ種などが存在する。ジャポニカ米は炊飯するとふっくらして粘りのある食感となるが、インディカ米はパサパサした食感となる。この違いはアミロース含量から説明できる。アミロース含量はジャポニカ米の約20% に対し、インディカ米では約25% である。老化しやすいアミロースは硬さに影響を与えるため、パサパサとした食感は主に高いアミロース含量に由来する4。ジャポニカ米とインディカ米に含まれるアミロペクチンの構造を比べると、前者の分岐鎖の鎖長が短くなっている(図 4)。これは、分岐鎖を伸長する酵素であるスターチシンターゼ(SS)アイソザイムIIa(SSIIa)の活性が、ジャポニカ米では弱いことに起因する3,4(「5.アミロペクチンおよびグリコーゲンの生合成に関わる酵素群」参照)。アミロペクチンの分岐鎖の鎖長は短いと老化しにくくなり、長いと老化しやすくなるため、アミロペクチンの構造も食感に影響を与えている。
グリコーゲンは、アミロペクチンと同様に α-1,4-グルカン直鎖および α-1,6-分岐点を介した分岐鎖から成るが、分岐構造に規則性は無く、分岐点が多く(割合は8–10%)分岐鎖が短い(「3. 分岐 α-グルカンの構造解析法」参照)ため、結晶性を示さずに水溶性となる1(図 1D)。また、らせん構造を形成しないため、ヨウ素で染色されない。
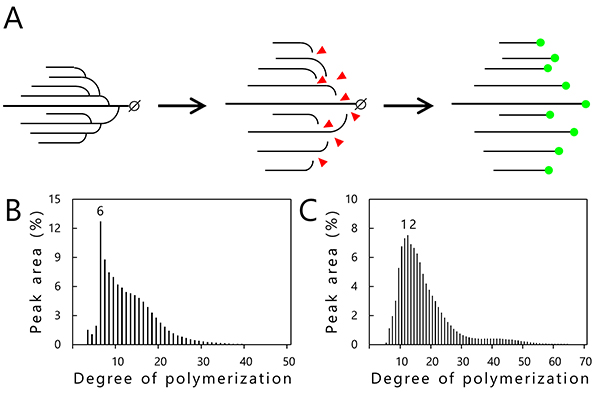
アミロペクチンとグリコーゲンの構造解析には、鎖長分布解析法が用いられている5。この手法では、枝切り酵素(イソアミラーゼ)で分岐点(α-1,6-結合)を特異的に加水分解し(図 5A)、α-1,4-グルカン直鎖とする。この処理で生じた還元末端を蛍光(APTS; 8-アミノピレン-1,3,6-トリスルホン酸三ナトリウム塩)でラベル化し(図 5A)、キャピラリー電気泳動装置を用いてグルカン鎖の鎖長ごとに分離すると、横軸および縦軸にそれぞれ含まれる分岐鎖の鎖長(グルコースの重合度; DP)および含まれる分岐鎖の本数を示すグラフが得られる5(図 5B, 5C)。市販のカキ由来グリコーゲンはDP6の分岐鎖を多く含み(図 5B)、イネ(ジャポニカ種日本晴)の胚乳から精製したアミロペクチンはDP10–13の分岐鎖を多く含む(図 5C)。これは、アミロペクチンのクラスターには、グルコース10–13残基から成る分岐鎖が多く含まれることを示している。鎖長分布解析法は、分岐鎖の鎖長と本数を知ることができるため構造解析の強力なツールであるが、分岐点の位置情報は得られない。また、らせん構造の組み方(図 1C)の情報も得られないので、平面モデル3(図 1B)を予想するに留まる。分岐 α-グルカンはグルコースのみから成るが、シンプルなゆえに構造解析が難しいという問題点がある。
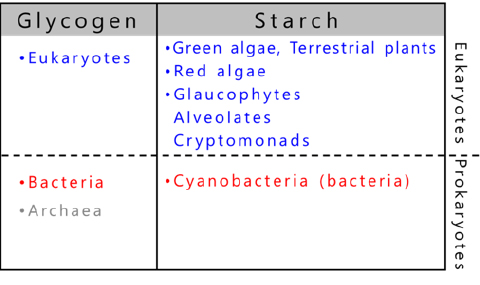
グリコーゲンを貯蔵する生物は、生物界の3つのドメインに広く分布しており6,7(図 6)、これらの生物はグリコーゲンをエネルギー源として利用している。それに対して澱粉は、光合成産物として一部の真核生物が貯蔵する6-9。澱粉を貯蔵する生物は、緑藻および緑藻から進化した陸上植物、紅藻、灰色藻、そして紅藻に由来するクリプト藻・アルベオラータに限定されている9(図 6)。原核生物であるシアノバクテリアは数千種存在すると言われており10、一般にグリコーゲンを貯蔵するが、ごく一部の種(約10種)は、例外的にアミロペクチン(シアノバクテリア澱粉)を貯蔵することが見出されている8,11(5. アミロペクチンおよびグリコーゲンの生合成に関わる酵素群」参照)。
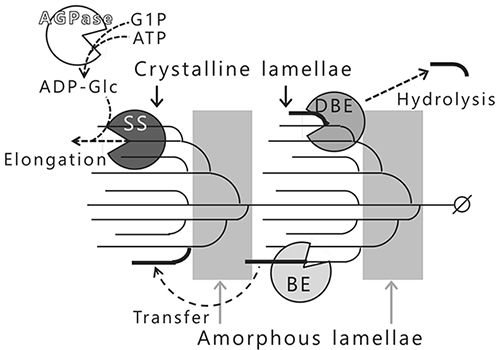
アミロペクチンの生合成には、主に4種類の酵素が関わっている3,4(図 7)。ADP-グルコースピロホスホリラーゼ(AGPase; EC 2.7.7.27)は、グルコース-1-リン酸とATPからADP-グルコース(ADP-Glc)を生産する。ADP-Glcはスターチシンターゼ(SS; EC 2.4.1.21)のドナー基質となり、SSはADP-Glc由来のグルコースをアクセプター基質であるα-1,4-グルカン鎖の非還元末端側に1残基ずつ転移する。この反応を繰り返すことで分岐鎖が伸長される(図 7)。枝作り酵素(BE; EC 2.4.1.18)はSSによってDP12以上にまで伸長された分岐鎖を切断し、分子内または分子間で転移することで新しい分岐点を形成する(図 7)。BEはまれにクリスタルラメラの不適切な位置に分岐点を形成するが、この分岐点は枝切り酵素(DBE, イソアミラーゼ(ISA; EC 3.2.1.68))によって特異的に加水分解されてアミロペクチンの構造が整えられ、結晶性を示すようになる(図 7)。陸上植物のアミロペクチンは、SS、BE、およびDBEが適材適所で作用することによって生合成される3,4,12。
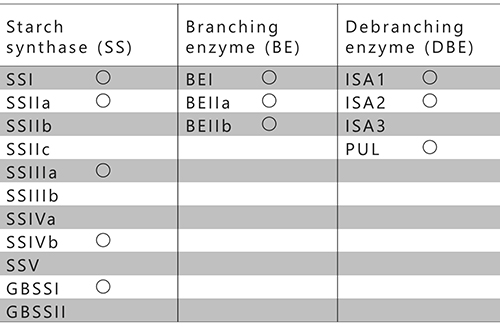
これまでに知られているイネ由来SS、BE、およびDBEのアイソザイムを表 1に示す。イネはSS、BE、およびDBEのアイソザイムを、それぞれ11種類、3種類、および4種類有している。これらは、貯蔵澱粉または同化澱粉の代謝に関わっている13(表 1)。DBEのうち、アミロペクチンの生合成に関わるアイソザイムはISA1およびISA2であり14,15、ISA316およびPUL17は分解に関わっている。ただしPULについては、合成にもわずかに関わることが分かっている17。藻類のアミロペクチン生合成系においても、AGPase、SS、BE、およびISAの機能は共通している。
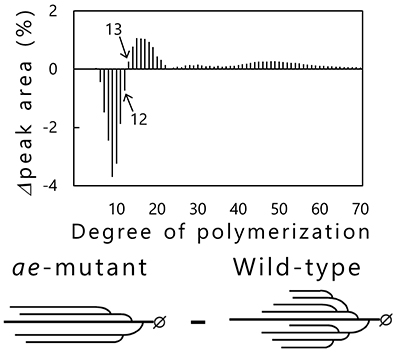
バクテリアのグリコーゲン生合成に関わる酵素群はアミロペクチン生合成に関わるものと共通しており、いずれもアミノ酸配列は類似している6,18。ただし、SSに対応する酵素はグリコーゲンシンターゼ(GS)と呼ばれ、バクテリア由来DBEはISAまたはGlgXと呼ばれる19。GSおよびBEはグリコーゲンの生合成に関わるが、DBEは分解に関わる点でアミロペクチンを蓄積する植物とは異なっている。グリコーゲンを蓄積する生物が持つ関連酵素のアイソザイム数は、植物と比べて少ないという特徴がある。たとえば大腸菌では、GS、BE、およびGlgXをそれぞれ1種のみ有する。シアノバクテリアは原核生物としてはめずらしく、アミロペクチンを蓄積する種も存在する8,11(「4. グリコーゲンおよび澱粉を貯蔵する生物の分布」参照)。アミロペクチン生産性シアノバクテリアは、植物と比べて関連酵素のアイソザイムが少ない8,20-22(表 2)。また、陸上植物で見出される酵素-酵素複合体(「6. 生合成酵素が澱粉の構造・性質に与える影響」参照)はこれまでに見つかっていない。これは、シアノバクテリアは地球上で最もシンプルなアミロペクチン生合成系を持っていることを示している。さらにアミロペクチン生産性シアノバクテリアは、アミノ酸配列がその他の生物とは異なる未解析のアイソザイムを複数持つ点で興味深い。シアノバクテリアは葉緑体の祖先生物であり、植物の澱粉代謝の多くはシアノバクテリアに由来している23,24。すなわち、シアノバクテリアのアミロペクチン生合成系を明らかにすれば、アミロペクチン生合成系はグリコーゲン生合成系からどのように進化したのか、明らかにできると期待される。
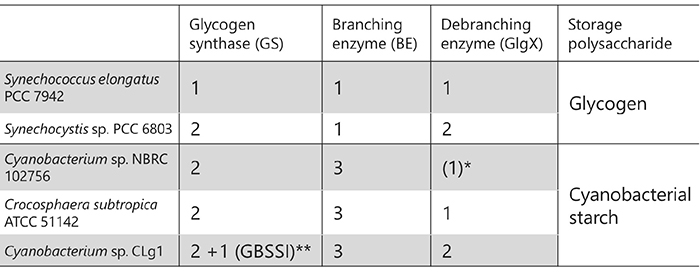
イネの胚乳で発現している澱粉生合成関連酵素について、個別の酵素を欠損させた変異体が胚乳に蓄積する澱粉の表現型を調べることで、酵素の機能が解析されている4,25(表 1)。たとえばBEIIbを欠損した変異体はamylose-extender(ae)と呼ばれ26、蓄積される澱粉は糊化しにくい、および難消化性が増加するなどの性質を示す27,28。これは、アミロペクチンの分岐鎖の本数が減少し鎖長が長くなるためである(図 8)。ae変異体の鎖長分布から野生型(ジャポニカ種金南風)の鎖長分布を引き算した差分グラフを作成すると、ae変異体は短い分岐鎖(DP < 13)が減少し、長い分岐鎖(DP ≥ 13)が増加している(図 8)。この実験からBEIIbの役割は、長い分岐鎖の切断および短い分岐鎖の生産であることが証明されている26。胚乳で発現しているその他のアイソザイムについても、同様の解析方法で役割が調べられている4,25。
これまでの研究からアミロペクチンのアモルファスラメラとクリスタルラメラの繰り返し構造は、複数のアイソザイムの共同作業で作られることがわかっており、アミロペクチン生合成のモデルが報告されている3,4,12。最近、陸上植物は関連酵素同士が相互作用し、酵素-酵素複合体を形成することがわかってきた25,29-31。たとえばイネ(日本晴)では、SSI、SSIIa、およびBEIIbが相互作用して複合体として存在することが証明されている25,30。澱粉生合成における酵素複合体の役割はまだわかっていないが、下記の未解決の問題点に関わっている可能性がある。
これまでに、生合成に関わる個別の酵素のin vivoとin vitroでの機能はわかっているが、アミロペクチンの構造を制御する仕組みはまだわかっていない。未解決の問題点を以下に示す。
これらを解決できれば、アミロペクチンの物性を意図的にコントロールできるようになり、新規澱粉素材としてバイオプラスチックなどへ応用できる可能性がある。
真核生物および原核生物に分布しているBEをアミノ酸配列の類似性に基づいて分類すると、いずれも糖加水分解酵素(GH)のファミリー13(GH13)に分類される32。真核生物由来BEおよび原核生物由来BEはアミノ酸配列が微妙に異なっており、それぞれGH13サブファミリー8(GH13_8)およびGH13サブファミリー9(GH13_9)に属し33、系統樹上で異なる位置に見出される(一部例外あり)6,7(図 9)。真核生物および原核生物由来BEは、carbohydrate-binding module (CBM) family 48 (CBM48)、ドメインA(触媒ドメイン)、およびドメインCを普遍的に有している6,7(図 10)。真核生物由来BEはN末端側に長い α-へリックスを持つのに対し、原核生物由来BEはN末端側にドメインNを持つものおよび持たないものが存在し、それぞれ group 1およびgroup 2と呼ばれる34。多くのシアノバクテリアは、group 1に属するBE1を有している(図 9, 図 10)。アミロペクチン生産性シアノバクテリアは3種のアイソザイム(BE1、BE2、およびBE3)を持ち、BE2(BE1とのアミノ酸配列相同性は約70%)およびBE3(BE1とのアミノ酸配列相同性は約20%)はgroup 2に属している(図 9, 図 10)。
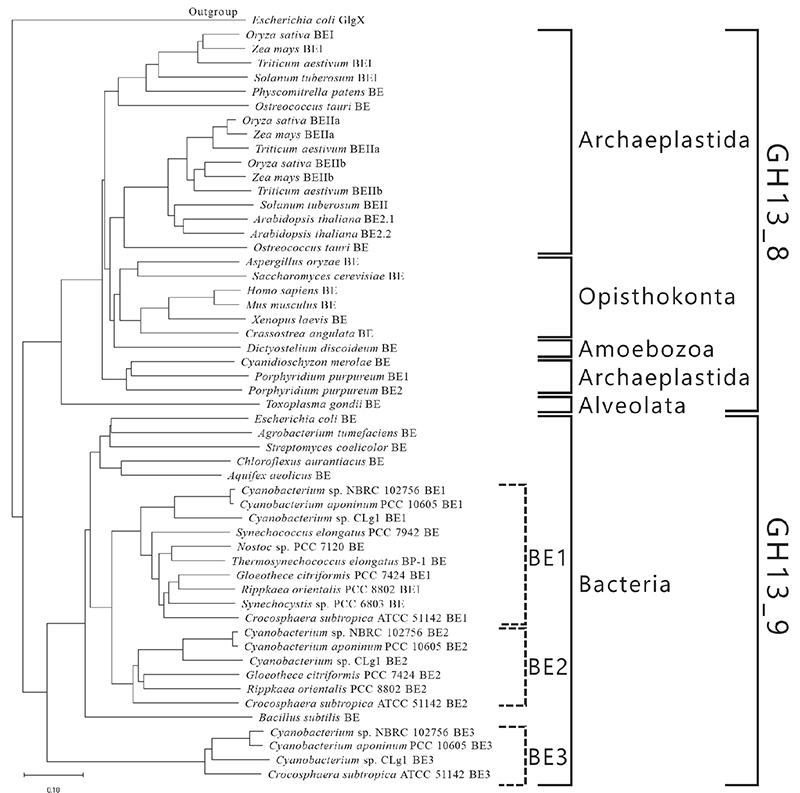

アミロペクチンの分岐鎖は、結晶性、粘性、老化のしやすさ、および食味などの物性に影響を及ぼす。著者らはBEが生産する分岐鎖の長さを制御することを目的として、シアノバクテリア由来BEの研究を進めているので、紹介したい。アミロペクチン生産性シアノバクテリアCrocosphaera subtropica ATCC 51142株由来cceBE1、cceBE2、およびcceBE3の精製リコンビナント酵素を調製し、生産される分岐鎖の数と鎖長(鎖長分布)を調べた21。cceBE1(図 11A)およびcceBE2(図 11B)は、DP12以上から成る分岐鎖を基質として切断し、DP6および7から成る分岐鎖(短鎖)を特異的に生産する。それに対して、cceBE3(図 11C)は特異的に生産する分岐鎖は無く、短鎖(DP5–11)およびわずかに長鎖(DP30–35)を生産する。イネ由来BEについてもリコンビナント酵素の特性が調べられており12、全てのBEの特性はイネ由来BEI、BEIIa、およびBEIIbのいずれかのタイプに分類できると言われている35,36。イネ由来BEI、BEIIa、およびBEIIbはDP12以上の分岐鎖を基質として、糖転移反応を触媒する。イネBEIIaはDP6および7の短鎖を主に生産し、DP8–15の分岐鎖も少し生産する。イネBEIIbはもっぱらDP6および7の短鎖を生産するが、ごくわずかにDP8–15の分岐鎖も生産する。イネBEIは短鎖(DP6–12)および長鎖(DP27–38)をわずかに生産する12。シアノバクテリア由来BE1およびBE2の特性はイネ由来BEIIbと類似していることからBEIIbタイプに、BE3はイネ由来BEIと類似していることからBEIタイプに分類できる20,21。原核生物由来BEは真核生物由来BEと独立して分化しているが、両者の特性は共通している。
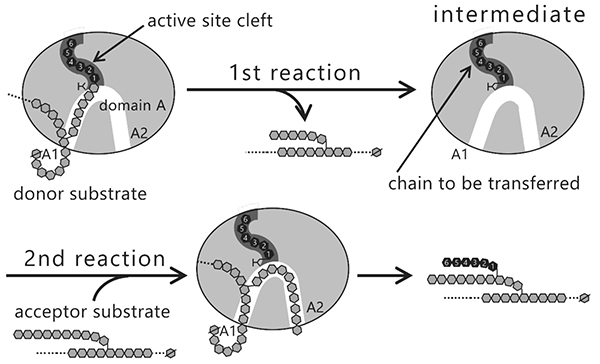
ここで、生産される分岐鎖の鎖長はBEアイソザイムによってなぜ変わるのかという疑問が生じる。BEが触媒する反応は、グリコシレーション反応(図 12, 1st reaction)とデグリコシレーション反応(図 12, 2nd reaction)の2段階から成る7,22,37,38。1st reactionではグルカン鎖のα-1,4-結合が切断されて転移鎖となり、BEの活性部位クレフトに転移鎖が結合した中間体が形成される。2nd reactionでは、BEが転移鎖をα-1,6-結合で転移して新しい分岐点を形成する。この中間体を作る仕組みは、アミラーゼやその他の糖質関連酵素でも見られる32,39。この反応の仕組みに基づくと、転移鎖の鎖長は活性部位クレフトが制御していると考えるのが合理的である。
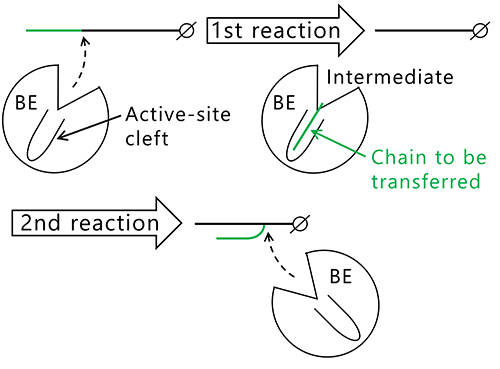
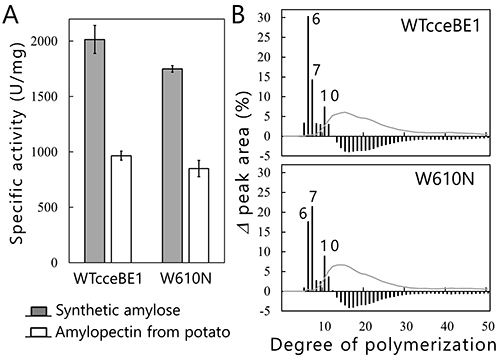
BEの結晶構造からこの仮説を検証したい。これまでに、大腸菌由来BE40-42(図 13A)、結核菌由来BE37(図 13B)、イネ由来BEI43,44(図 13C)、およびヒト由来BE45(図 13D)の結晶構造が報告されているが、活性部位クレフトに糖鎖が結合した状態の結晶構造はこれまでに報告されていなかった。著者らはこの問題を解決するため、cceBE1の結晶構造解析に挑戦し、マルトオリゴ糖が結合した状態の結晶構造の解明に成功した22。野生型cceBE1(WTcceBE1)の活性部位クレフトには、DP6の転移鎖がぴったりと結合していた(図 14A)。次に活性部位クレフトが酵素特性に与える影響を調べるため、転移鎖の非還元末端側の溝を形成しているアミノ酸残基(図 14A, 黒丸)を他の残基に置換した変異体の構築および解析を行った。その結果、WTcceBE1と同等の比活性を維持しているW610N変異体を創出した22(図 15A)。WTcceBE1は主にDP6から成る分岐鎖を生産するが(図 11A, 図 15B)、W610NではDP 6の生産割合が減少し、DP 7を主に生産するように変化していた(図 15B)。このことからW610Nの活性部位クレフトの非還元末端側(図 14A, 黒丸)には、DP7の転移鎖が結合するスペースが生じたと予想された。そこで、W610N にDP7の転移鎖(G7)が結合した状態の結晶構造を決定したところ、G7は活性部位クレフトにぴったり結合していた22(図 14B)。これらの結果は、活性部位クレフトが転移鎖の鎖長を制御していることを強く示唆している。
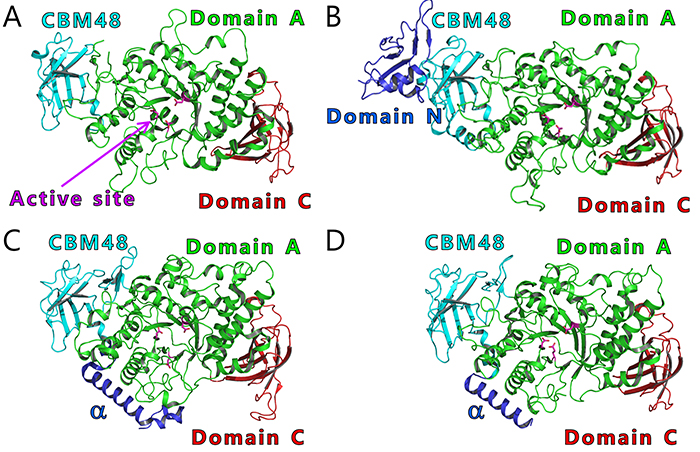
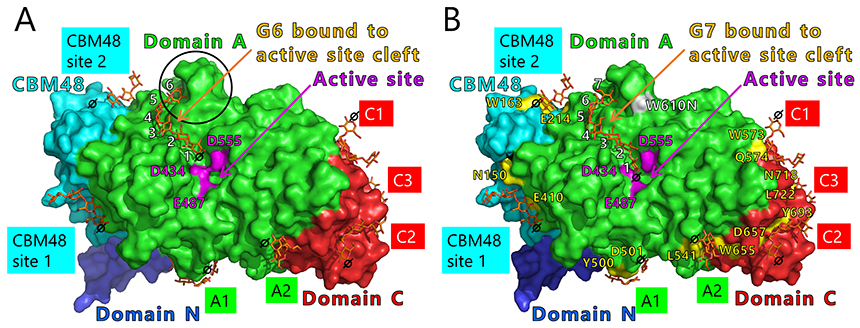
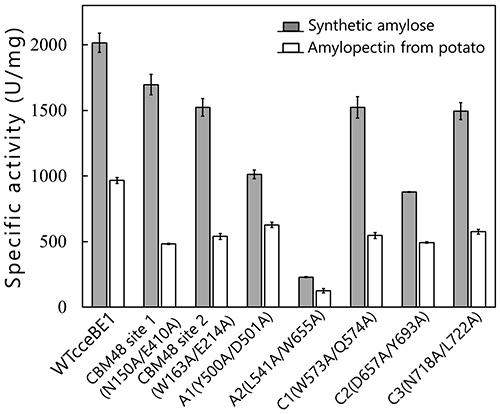
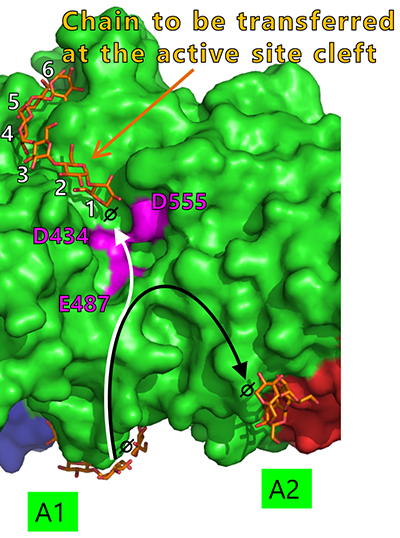
WTcceBE1およびW610Nの酵素表面には、マルトオリゴ糖が7ヶ所に結合していた(図 14)。これらのうちCBM48の2ヶ所は、CBM48 site 1およびCBM48 site2とした46。それ以外の5ヶ所は酵素表面糖鎖結合部位(SBS)47,48とみなし、A1、A2、C1、C2、およびC3とした46(図 14)。これらの糖鎖と相互作用しているアミノ酸残基(図14B, 黄色)をアラニンに置換した変異体の解析を行ったところ、A1(Y500A/D501A)およびA2(L541A/W655A)変異体はWTcceBE1と比べて比活性が低下したことから22,46(図 16)、BEの触媒反応に重要であることが示された。A1、A2、および活性部位クレフトに結合している糖鎖は、A1から活性部位クレフトまで(図 17, 白矢印)、およびA1からA2まで矛盾なく繋がる(図 17, 黒矢印)。このことから、ドナー基質とアクセプター基質は、それぞれ白矢印および黒矢印(図 17)のように結合すると考えられた。これまで解説してきた内容に基づいて、BEの反応機構のモデルを考案した22。まず、ドナー基質がA1から活性部位クレフトにかけて結合し、1st reactionが起こる(図 18, 上段)。ドナー基質のα-1,4-結合は切断され、活性部位クレフトに結合した転移鎖は中間体として残り、それ以外の糖鎖は遊離する(図 18, 上段)。次いでアクセプター基質がA1からA2にかけて結合して2nd reactionが起こり、α-1,6-結合から成る新しい分岐点が生産される(図 18, 下段)。この反応モデルは、実際に生産される多糖の分岐構造を矛盾なく説明できる。これまでにBEの反応モデルは報告されていなかったが、著者らはcceBE1の結晶構造解析の成果から報告するに至った22。
本稿では、BEが生産する分岐鎖の鎖長を制御する仕組みを解明した。澱粉は食料のみならずプラスチックの代替素材などとしても利用可能である。澱粉の利用を通して、人類が抱えている食糧問題および地球温暖化問題の解決に貢献できる可能性がある。そのためには澱粉生合成の仕組みを明らかにして構造・性質を制御する必要があるが、未解決の課題(「6. 生合成酵素が澱粉の構造・性質に与える影響」参照)が残されているため、さらに研究を続けて解決していきたい。
本稿(邦文・英文)は、第14回多糖の未来フォーラム2020で発表された内容を文章化したものである。本稿で紹介したBEの構造機能解析の研究成果は、島津科学技術振興財団研究助成、日本応用酵素協会酵素研究助成金、およびKAKENHI(25440193、15K18685、16K07467、18K06135)のご支援の下、多くの学生さんおよび研究者のご協力によって成し遂げたものである。心より感謝申し上げますとともに、今後ともご指導ご支援を賜りますようお願い申し上げます。