
氏名:加藤 紀彦
京都大学大学院生命科学研究科准教授
京都大学農学部生物機能科学科卒業(2001年)。山本憲二教授の指導の下、2007年に京都大学大学院生命科学研究科にて博士号(生命科学)を取得。University of Georgia、Complex Carbohydrate Research Center、Michael Tiemeyer教授の下2013年までポスドク研究員。2013年石川県立大学生物資源工学研究所助教、2015年京都大学大学院生命科学研究科助教を経て2023年より現職。研究テーマ:ヒト腸内細菌と宿主由来複合糖質の相互作用。

氏名:片山 高嶺
京都大学大学院生命科学研究科教授
京都大学農学部食品工学科を卒業後、同大学院に進学し、熊谷英彦先生の下で学位を取得。応用微生物学分野を専門とする。同研究室で3年間博士研究員として在籍した後に、京都大学大学院生命科学研究科統合生命科学専攻・山本憲二教授研究室の助手に着任し、糖質関連酵素に関する研究をスタートさせる。ビフィズス菌がヒト由来の糖質に作用する酵素を有していることを見出し、ヒトと腸内細菌の共生に着目する。母乳栄養児におけるビフィズスフローラ形成機構の解明に貢献した。
略語
CBM, carbohydrate-binding module; Fuc, fucose; Gal, galactose; GalN, galactosamine; GalNAc, N-acetylgalactosamine; GH, glycoside hydrolase; GlcNAc, N-acetylglucosamine; GlcNAc-6S, 6-O-sulfated N-acetylglucosamine; LacNAc, N-acetyllactosamine; Neu5Ac, N-acetylneuraminic acid.
特定の腸内細菌はヒト腸管への適応戦略としてムチン分解酵素を産生する。近年、それら酵素群の解析が進み、ムチンの複雑な構造に対応した多様な酵素レパートリーが存在することが明らかとなってきた。またそれら酵素に関する情報は各種データベースにまとめられ、腸管内での分解・代謝経路の包括的な理解を試みる上で有用である。本稿ではこれまで明らかにされた腸内細菌由来のムチン分解関連酵素について焦点をあて、酵素の特徴から推定されるムチン分解経路について考察する。
腸管粘膜組織は消化・吸収の場であると同時に、特に大腸においては腸内細菌との共生の場でもある。消化管上皮から分泌される多量のムチンは、粘液層を形成して組織を保護し、細菌やウイルスなどを絡め取り体外へと排出する役割を持つ1。一方で病原性菌の一部はこのムチン粘液層を分解・浸食して宿主体内へ侵入する。ところが非病原性の常在性細菌やプロバイオティクス(いわゆる善玉菌)においてもムチン分解活性を有する菌が存在し、ムチンの資化・利用によって腸管内で増殖することが明らかとなってきた2。さらにはムチン分解産物である遊離糖は細菌叢内で共有・代謝され、短鎖脂肪酸など有益な代謝物に変換されることから、ムチン資化は宿主の健康やバランスの取れた菌叢(eubiosis)の維持にとっても重要であると考えられている。従って、ムチン分解の宿主に対する影響は、病理面および共生面の両面から考える必要がある。ただし、それらを包括的に理解する上では、ムチン分解活性を有する各菌のムチン分解経路について知ることが重要である。本稿では主要なムチン資化性腸内細菌が有する酵素レパートリーについて紹介するとともに、推定される各菌のムチン分解経路について考察したい。
ムチンには分泌型のゲル形成ムチンと膜貫通ドメインを有する膜結合型ムチンの大きく2つが存在し、両方に共通してプロリン、トレオニン、セリンに富む領域(ムチンドメイン)が存在する。このムチンドメイン内のトレオニンあるいはセリン残基の水酸基に、GalNAcがα-位で結合するO-結合型糖鎖(ムチン型糖鎖、O-glycan)が密集して付加されることがムチンの特徴である。健常人の腸管に見られるO-glycanはコア1~4構造を有し、さらにtype-1/2 LacNAc構造による伸長、そしてフコース、シアル酸、硫酸基、およびアセチル基などによる修飾が施され、極めて不均質で多様な構造を取り、さまざまな末端糖鎖抗原を提示する(図 1)。O-Glycan生合成に関する詳細は他文献を参照いただきたい3。
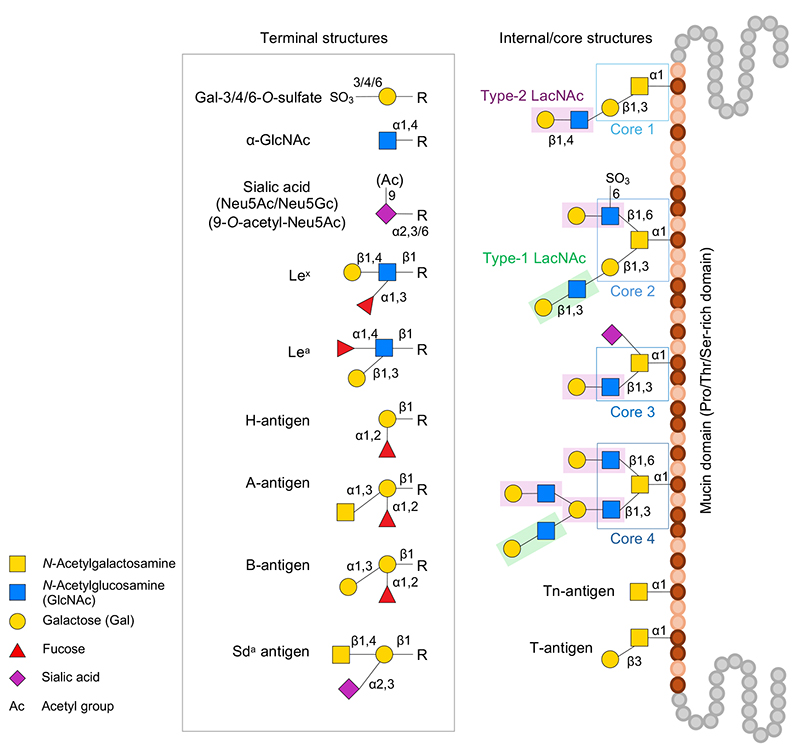
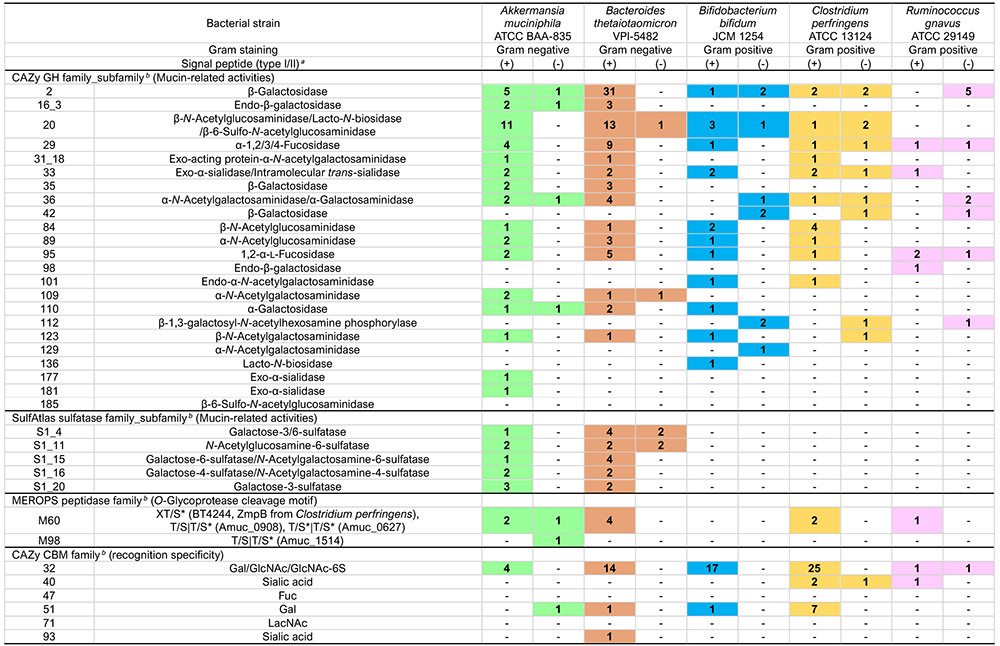
糖質加水分解酵素(glycoside hydrolase: GH、EC3.2.1.-)は、CAZyデータベース(http://www.cazy.org/)4において187のファミリーに分類されている(2023年12月時点)。そのうち少なくとも23のGHファミリー、サブファミリーに実験的にムチン糖鎖分解への関与が確認されるか、あるいは関与の可能性が考えられている(表 1)。
糖鎖末端修飾の分解に関わるGH酵素は以下のものがある。GH95 1,2-α-ʟ-fucosidaseはH抗原(Fucα1-2Galβ1-R)に、またGH29 1,3/4-α-ʟ-fucosidaseはルイス型抗原に含まれるFucの遊離に関わる5-8。シアル酸の遊離にはGH33およびGH181 exo-α-sialidaseが関与する8-10。GH177 exo-α-sialidaseは歯周病菌Tannerella forsythensisにおいて同定され11、Akkermansia属細菌などからも見出されているがムチンに対する活性の有無は未確認である。Ruminococcus gnavus由来GH33酵素はintramolecular trans-sialidase活性を有しており、α-2,3結合のNeu5Acに特異的に作用して2,7-anhydro-Neu5Acを生成・遊離する12,13。α-結合した末端GlcNAc残基にはGH89のα-N-acetylglucosaminidaseが作用する14,15。A型あるいはB型血液型抗原(GalNAcα1-3(Fucα1-2)Galβ1-R、Galα1-3(Fucα1-2)Galβ1-R)にはそれぞれGH36/GH109 α-N-acetylgalactosaminidaseおよびGH110 α-galactosidaseが作用してH抗原に変換する16,17。A型抗原のGalNAc残基にGalNAc deacetylaseが作用してGalNに変換後に、GH36 α-galactosaminidaseが作用して分解する経路も知られている18。またGH98 endo-β-galactosidaseはA/B型血液型抗原3糖を遊離する19。
Type-2 LacNAc(Galβ1-4GlcNAcβ1-R)の分解にはGH2 β-galactosidaseおよびGH20/GH84 β-N-acetylglucosaminidaseが関与する20,21。Type-1 LacNAc(Galβ1-3GlcNAcβ1-R)の分解にはGH35 β-galactosidaseの関与が考えられているが22、それ以外にGH20/GH136 lacto-N-biosidaseも関与する。後者の場合はlacto-N-biose I(LNB)の2糖が遊離する23–25。それ以外にもGH42 β-galactosidaseは細胞内でtype-1/2 LacNAc構造の分解に関与する可能性がある26,27。GH16_3 endo-β-galactosidaseはO-glycan内部のβ-Gal結合に作用してオリゴ糖を遊離するが、その反応に先立って糖鎖末端のシアル酸除去が必要とされる28。
コア1(T抗原、Galβ1-3GalNAcα1-Ser/Thr)はtype-1 LacNAc の場合と同様にGH35 β-galactosidaseが作用してTn抗原(GalNAcα1-Ser/Thr)に変換される可能性が考えられる22。またGH101 endo-α-N-acetylgalactosaminidaseはT抗原からGalβ1-3GalNAc(galacto-N-biose、GNB)の2糖を遊離する29,30。コア2(Galβ1-3(GlcNAcβ1-6)GalNAcα1-Ser/Thr)にはGH84 β-N-acetylglucosaminidaseが作用することでコア1に変換される21。一方で、6位硫酸化GlcNAc(GlcNAc-6S)を含むコア2(Galβ1-3(6-sulfo-GlcNAcβ1-6)GalNAcα1-Ser/Thr)にはGH20 6-sulfo-β-N-acetylglucosaminidase (sulfoglycosidase)が作用してコア1に変換する31。GH185 6-sulfo-β-N-acetylglucosaminidaseもムチン糖鎖中のGlcNAc-6S残基の分解に関与する可能性がある32。コア3(GlcNAcβ1-3GalNAcα1-Ser/Thr)にはGH20 β-N-acetylglucosaminidaseが作用してTn抗原に変換する21。Tn抗原にはGH31_18/GH129 α-N-acetylgalactosaminidaseが作用してGalNAcを遊離する33,34。
GHファミリーはアミノ酸配列の相同性によって分類されているが、同じファミリーであっても特異性の異なる複数の活性を含むことが多く、また未知の基質特異性を示す可能性もある。したがって上記のGHがゲノム上に存在しているからと言って必ずしもムチンに作用するとは限らないことに留意する必要がある。
Sulfatase(sulfohydrolase、EC3.1.6.-)のデータベースとしてSulfAtlas(https://sulfatlas.sb-roscoff.fr/)35が整備されている。それによるとsulfataseはS1~S4の4つのファミリーと、多くのサブファミリーに分類される。特にS1では110のサブファミリーが存在する(S1_Xと表記)。硫酸化糖に作用するcarbohydrate sulfatase活性を有するものはS1ファミリーに属する。Sulfataseの活性中心はCysもしくはSer残基が翻訳共役的に修飾されることで生成するCα-formylglycine残基であるが36、この反応は嫌気性細菌においてはanaerobic sulfatase-maturating enzyme(anSME)によって触媒される。
ムチンの硫酸基修飾はGalの3、4、6位、あるいはGlcNAcの6位、またNeu5Acの8位37などで検出される。腸内細菌由来sulfataseのうち、特にBacteroides thetaiotaomicron由来酵素に関してMartensらによって多くの情報が提供された38,39。B. thetaiotaomicronは実に28個ものS1 sulfatase遺伝子を保有し、それらはムチンやグリコサミノグリカンなどの多糖利用関連遺伝子クラスター(polysaccharide utilization loci、PUL)中に存在する。ムチン関連PULとしてSusC/SusD(細菌が多糖の分解産物を菌体内に取り込むシステム)やGHなどともにクラスターを形成するものには、S1_4、S1_11、S1_15、S1_16、S1_20がある40。
硫酸化Galに作用するものにはS1_4、S1_15、S1_16、S1_20があるが、表 1に示すようにS1_4およびS1_20は3位硫酸化Galに作用するgalactose-3-sulfatase活性を示し、S1_15は6位硫酸化Galあるいは6位硫酸化GalNAcに対するgalactose-6-sulfataseおよびN-acetylgalactosamine-6-sulfatase活性を示す。S1_16は4位硫酸化Gal(GalNAc)に対するgalactose-4-sulfatase、N-acetylgalactosamine-4-sulfatase活性を示す。6位硫酸化GlcNAc(GlcNAc-6S)に対しては、S1_11が特異性を示す。またBifidobacterium breve由来のS1_53酵素もGlcNAc-6S代謝に関わるとする報告がある41。なお硫酸化シアル酸に作用するsulfataseは筆者の知るところでは未だ同定されていない。
O-GlycoproteaseとはO-glycanを持つペプチドに作用するprotease(peptidase)のことである。OgpAやCpaAなどのO-glycoproteaseはO-glycanを持つ糖タンパク質に対して広い特異性を示すが、本稿で注目したいのはムチンドメインを有する糖タンパク質に特異的なO-glycoproteaseであり、これらはムチンドメイン構造の認識や後述のcarbohydrate-binding module(CBM)の保有などの特徴を有することからmucinaseとも呼称される42。病原性菌のみならず常在性の腸内細菌もmucinaseを分泌しムチンポリペプチドを分解する。これら酵素の多くは、ペプチダーゼとその阻害剤のデータベースであるMEROPS peptidase family43のいくつか(M60, M66, M98など)に分類される細胞外亜鉛要求性メタロプロテアーゼであり、M60-likeファミリー(Pfam 13402)を構成する44。M60-likeファミリーはドメイン中にHEXXHモチーフを共通して有する。モチーフ中の二つのヒスチジン残基は亜鉛イオンの結合に関与し、グルタミン酸残基は触媒残基である。M60-likeファミリー以外ではserine proteaseなどにもO-glycoprotease活性が見出されている。
O-Glycoproteaseの基質ポケットが認識する糖鎖構造は酵素によって異なるが、 (sialyl)Tや(sialyl)Tn抗原などの比較的短くトリミングされたO-glycanを認識するものが多いため45、GHによる糖鎖トリミングがO-glycoproteaseの分解を促進すると推察される。M60ファミリーのいくつかは病原性と関連することが知られており46、これはムチン層の骨格をなすムチンポリペプチド鎖を直接分解することで、病原菌が上皮細胞へ侵入しやすくなるためと考えられる。
近年、O-glycoproteaseの応用展開が急速に広がり、ムチンドメイン含有糖タンパク質の詳細な構造解析47やムチン染色45などへの解析ツールとしての利用が大きな注目を集めている。
糖鎖結合モジュール(carbohydrate-binding module, CBM)は糖鎖構造の認識に関わる附属ドメインであり、CAZyデータベースにおいて現在100のファミリーに分類されている。ムチン糖鎖の認識に関わるものとしてCBM32/CBM40/CBM47/CBM51/CBM71/CBM93が考えられる。中でもCBM32はGal/GalNAc/GlcNAc/GlcNAc-6Sの認識に関与し、多くのGHやO-glycoprotease中に見出される。CBMによるムチン糖鎖への結合は酵素の直接的なムチンへの接触を可能とするため、結果的にムチン分解効率を向上させる効果を有する31。
その他のムチン分解に関連する酵素としては、9-O-アセチル化シアル酸の分解に働くsialate-O-acetylesteraseや、LNBやGNBを加リン酸分解するLNB/GNB phosphorylase(GH112)などが知られている48。Sialate-O-acetylesteraseはsialidaseとのマルチドメイン酵素として見出される場合もある9。なお既知LNB/GNB phosphorylaseは細胞内酵素であるため直接ムチンに作用する例は知られていない。
細菌の種や株によって上記酵素群の保持パターンは異なるため、ムチン分解のパターンも一様ではない。また上述した通り、同じファミリーの酵素を保持する場合でも活性や特異性が同じかどうかは実験的に確認する必要がある。さらに、同様の活性を示す場合であっても、それが細胞外で作用する場合と細胞外小胞の積み荷タンパク質として輸送される場合、あるいはペリプラズム空間やサイトプラズムで作用する場合などでは分解経路や他菌への影響は大きく異なることが予想される。したがって各菌のムチン分解経路を考える上では酵素の種類だけではなく各酵素の局在性も考慮に入れる必要がある。
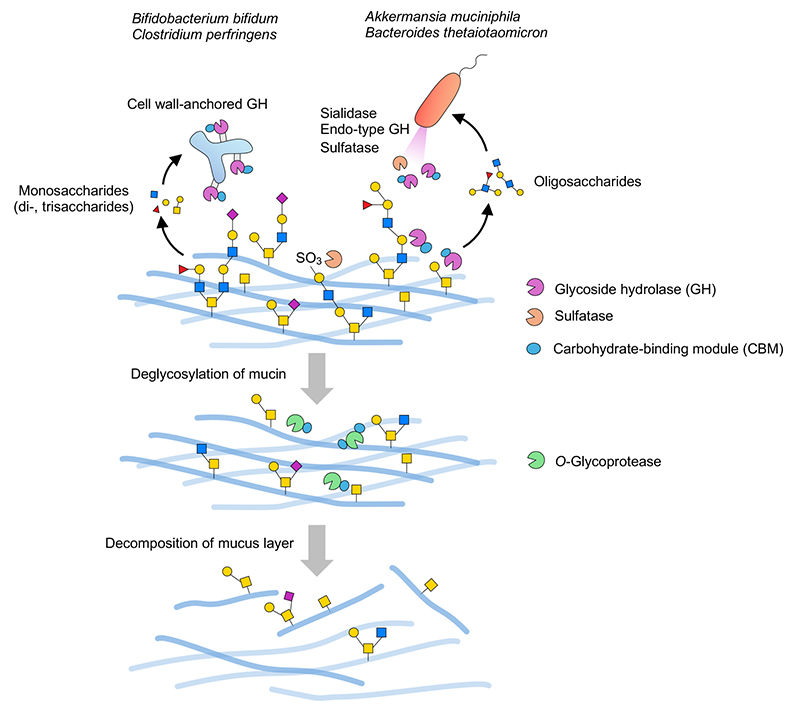
グラム陰性菌であるBacteroides属細菌などでは上述のように多くのPULを有することが知られている。植物多糖利用経路において多くの場合では、エンド型分泌酵素が細胞外で多糖をオリゴ糖に分解し、オリゴ糖はSusC/SusD取り込み系によってペリプラズム空間へと輸送される。ペリプラズム空間にはオリゴ糖分解酵素が待ち構えており、そこで単糖レベルに分解される。最終的に単糖はサイトプラズムに輸送されて資化される。本システムはムチン糖鎖においても同様と考えられる。Luisらの総説40やPULデータベース(PULDB)4などによるとB. thetaiotaomicronのムチンPULにはS1_20/S1_11等のsulfataseに加えてGH2/GH16_3/GH20/GH29/GH95などがコードされている。おそらく、分泌型GH33 sialidase(BT0455)28,49あるいは外膜あるいは外膜由来小胞局在性のリポタンパク質として予想されるS1_20 galactose-3-sulfatase(BT1636)38が最初に細胞外でムチンに直接作用し、それぞれシアル酸残基や硫酸基といった酸性の末端修飾残基が除去され、次いでGH16_3 endo-β-galactosidaseなどの細胞外エンド型酵素が作用してオリゴ糖を産生すると推測される28。オリゴ糖は外膜上のSusC/SusDによってペリプラズム空間へと輸送され、ペリプラズム局在性のsulfataseやGHによってさらに細かく分解される40。ムチン由来オリゴ糖をペリプラズムに取り込むことは、これらの糖を効率よく資化する上で重要と思われる。GH16_3による作用を受けたムチン糖タンパク質は、コア構造やそれに近い短い糖鎖構造を露出させると考えられるため、O-glycoproteaseの良い基質となりうるであろう(図 2)。
一方で、ペリプラズム空間のない一部のグラム陽性菌(BifidobacteriumやClostridium)は細胞壁アンカー型のGHを有している。これらのGHは多くの場合一旦膜タンパク質として生合成されたのちにsortase依存的経路によってペプチドグリカン層上に共有結合によって固定化される50。これら細胞表層局在性のGHはムチン糖鎖を末端から逐次分解することで単糖や短いオリゴ糖を環境中に遊離し(図 2)、利用可能な糖は菌体内に取り込まれる。菌体の表層に酵素を提示するため、効率よくムチンを分解するためには菌体自身がムチンに接着する必要があると考えられる。この点で、細胞壁アンカー型GH酵素中に多くのCBMが存在することは注目に値する。これらのCBMは基質との結合活性を示し、触媒ドメインの作用性向上に重要な役割を果たしている31。ムチンは巨大分子であり、液中での分散性は高くない。したがって基質と酵素の会合効率を高めるためのCBMによるムチン上の基質の捕捉が必須となると考えられる。なおBacteroidesやAkkermansia由来の酵素中にもCBMが見られることがあるが、細胞外酵素としてムチンに直接作用する酵素がCBMを持つことが多いようである。細胞外小胞の積み荷タンパク質として同定されているGHによるムチン分解メカニズムに関しては今後の解析が待たれる。
ところで、ムチン資化性細菌ゲノム情報をもとに各CBMファミリーの種類や数が各GHファミリーの有無とどのように関連するかを統計的に調べたところ、興味深いことにGH16_3 endo-β-galactosidaseの有無がCBMの保持性に最も大きな影響を与えている可能性が示された31。GH16_3を持たないグラム陽性菌のBifidobacterium bifidumとClostridium perfringensはCBM保持率の高いものが多く、一方でGH16_3を有するグラム陰性菌ではCBM保持率が低い。エンド型酵素で切り出されたオリゴ糖は液中での分散性が高いためにCBMがなくとも酵素との会合効率は高く、この関係性は理にかなっている。このことから腸内細菌によるムチン分解経路としてCBM依存型とエンド型酵素依存型の2つの分解様式の存在が推定された。興味深いことにC. perfringensの一部の株はエンド型酵素依存的経路を持つことが推察され、同一菌種内でもムチン分解様式が異なる場合もあった。またグラム陽性菌であるRuminococcus gnavusはCBMの保持率も低くGH16_3も持たないため、上記2つの経路とも異なる様式でムチンを分解するのかも知れない。あるいは、そもそもムチン分解酵素の数・種類も比較的少ないため、限定的なムチン分解活性しか示さない菌である可能性も考えられる。
最後に、我々がこれまで注目してきたビフィズス菌Bifidobacterium bifidumのムチン糖鎖分解機序について概説する。本菌はプロバイオティクスとしても知られ、成人のみならず乳児の腸管からも高頻度に検出される。また、ムチン糖鎖と構造類似性を示すヒトミルクオリゴ糖の分解・代謝も行うことが知られている51。
B. bifidumのムチン分解のほとんどのステップは、上述の細胞壁アンカー型GHによって細胞外で行われる。GH95 1,2-α-ʟ-fucosidase(AfcA)5、GH29 1,3/4-α-ʟ-fucosidase(AfcB)6 によってH抗原やルイス型抗原からFucが遊離され、GH33 sialidase(SiaBb19,52、SiaBb210,52,53)によってシアル酸が遊離される。SiaBb1はGH33ドメインの他にsialate-O-acetylesterase活性を示すSGNH hydrolase domainも有するマルチドメイン酵素である9。さらにはGH123 β-N-acetylgalactosaminidaseとsialidaseドメインを有する別のマルチドメイン酵素(SiaBb3)も見出されているが、これはSda抗原(Neu5Acα2,3(GalNAcβ1,4)Galβ-R)の分解への関与が推定される(芦田ら、unpublished data)。胃ムチンなどに多いとされるGlcNAcがα1,4位で結合したα-GlcNAcキャッピングの遊離にはGH89 α-N-acetylglucosaminidase AgnBが作用する15。B型抗原に作用する酵素としてGH110 α-galactosidase AgaBb54を有する一方で、A型抗原に作用するGH109 α-N-acetylgalactosaminidaseは同定されておらず、GalNAc deacetylaseおよびGH36 α-galactosaminidaseのホモログも保有していない。またtype-2 LacNAcの分解にはGH2 β-galactosidase BbgIIIが関与し、さらにβ-GlcNAc残基の分解にはGH20 β-N-acetylglucosaminidase BbhI、GH84 β-N-acetylglucosaminidase BbhIVの関与の可能性が考えられる20,21。Type-1 LacNAcの分解にはGH20/GH136 lacto-N-biosidaseが作用してLNBの2糖を遊離する23,55,56。B. bifidumは硫酸化糖に対するsulfatase遺伝子を持たないが、β-GlcNAc-6S結合はGH20 sulfoglycosidase BbhIIによって分解され、硫酸化糖として遊離される31。硫酸化Galの分解に関与する酵素は未同定である。コア2/コア4構造に含まれるβ1,6-GlcNAc残基にはGH84 β-N-acetylglucosaminidase BbhIVが作用して、それぞれコア1とコア3に変換される21。コア3のβ1,3-GlcNAc残基にはGH20 β-N-acetylglucosaminidase BbhIが作用してTn抗原に変換される21。コア1構造(T抗原)にはGH101 endo-α-N-acetylgalactosaminidaseが作用してGNBの2糖を遊離すると考えられている。遊離したLNBやGNBはABCトランスポーターによってサイトプラズムに輸送され、GH112 GNB/LNB phosphorylase LnpA1あるいはLnpA2によってGal-1-phosphateとGalNAc/GlcNAcに加リン酸分解される48。GH129 α-N-acetylgalactosaminidase NagBbはサイトプラズムに局在しており、Tn抗原を有するSer/Thrあるいは糖ペプチドに作用してGalNAcを遊離する34。しかし、その基質であるTn抗原を有する糖アミノ酸・糖ペプチドが産生されて細胞内に輸送される機構については未解明である。
上記のようにB. bifidumはムチン糖鎖に見られる多様な糖鎖構造に対応したGHレパートリーを有し、ヒト腸管ムチン層環境に生息するために高度に適応している。面白いことに本菌はGH16の遺伝子断片をゲノム上に有している。しかしGH16の活性中心部分が欠失しているために、その遺伝子産物は酵素活性を示さないと考えられる。このことはムチン層に対する適応進化の歴史において本菌がGH16_3エンド酵素依存戦略ではなくCBM依存的ムチン分解戦略を選択した進化的痕跡かもしれない。
これまで述べてきたように、ムチン分解に関わる多くの酵素が報告され各種データベースに情報が整理されつつある。今後もさらなる酵素や新規活性・特異性の発見によって知見は拡大していくことだろう。そしてこれらの知識を基盤とした生物間相互作用のより深い理解、さらには世界で増え続ける腸関連疾患に対する有効な手立ての構築・開発へとつながることを期待したい。